Caracterización de cultivos neuronales de hipocampo murino como modelo de estudio de la señalización NRGs/ErbB4
Murine hippocampal neuronal culture characterization as a model for the study of NRGs/ErbB4 signaling
Luis Felipe Monge-Mora1, María Clara Soto-Bernardini2
Fecha de recepción: 22 de octubre de 2020
Fecha de aprobación: 3 de febrero de 2021
Monge-Mora, L.F; Soto-Bernardini, M.C. Caracterización de cultivos neuronales de hipocampo murino como modelo de estudio de la señalización NRGs/ErbB4. Tecnología en Marcha. Vol. 35-1. Enero-Marzo 2022. Pág 44-53. https://doi.org/10.18845/tm.v35i1.5429
https://doi.org/10.18845/tm.v35i1.5429
Palabras clave
Neuregulinas; ErbB4; neuronas primarias de hipocampo; esquizofrenia.
Resumen
Las neuregulinas (NRGs) son una familia de factores de crecimiento tipo epidérmico que actúan modulando diversas respuestas celulares a través de la interacción con los receptores ErbBs. Las NRG1 y NRG2 son las principales en el Sistema Nervioso Central (SNC) y su principal receptor en el cerebro es el ErbB4. La señalización NRG1/ErbB4 ha sido involucrada en procesos relevantes para la formación y mantenimiento del SNC como la migración de interneuronas, la mielinización, la neurotransmisión, la modulación de sinapsis, entre otras. Por otro lado, la NRG2 también se une y activa al receptor ErbB4, sin embargo, su señalización ha sido menos explorada. Existe evidencia de que alteraciones funcionales en estos módulos de señalización podrían provocar disfunciones en redes neuronales, de relevancia en el contexto de enfermedades neuropsiquiátricas. Por lo tanto, es de gran importancia contar con modelos in vitro para el estudio de su señalización, caracterizados en cuanto a la proporción de tipos de neuronas en las cuales las proteínas se expresan, y a la expresión de sus genes. En el presente trabajo se determinó una proporción aproximada de 85% de neuronas glutamatérgicas y 15% de interneuronas GABAérgicas en cultivos primarios de neuronas de hipocampo de ratones C57BL/6 preparados del día embrionario (E) 18, similar a lo que se ha reportado in vivo e in vitro. Además, se determinó la expresión de los genes Nrg1 tipo III, Nrg2 y ErbB4 para el día in vitro (DIV) 7, siendo la expresión de Nrg1 tipo III mayor que Nrg2, similar a lo reportado en el hipocampo in vivo. Esto sugiere que los cultivos pueden ser utilizados como modelos in vitro válidos para el estudio de la señalización de las NRGs.
Keywords
Neuregulins; ErbB4; hippocampal neurons; schizophrenia.
Abstract
Neuregulins (NRGs) comprise a family of EGF-like growth factors that modulate diverse cellular responses by interacting with their receptors ErbBs. In the Central Nervous System (CNS) the principal members include NRG1 and NRG2, and their principal receptor is ErbB4. The NRG1/ErbB4 signaling has been involved in relevant processes for the development and maintenance of the CNS such as interneuron migration, myelinization, neurotransmission, and synapse modulation. NRG2 can also bind to ErbB4 and activate it: however, the NRG2/ErbB4 signaling remains less studied. There is evidence that functional disturbances in these signaling modules could lead to neuronal networks dysfunction, of importance in the context of neuropsychiatric diseases. Therefore, a characterized in vitro model in terms of the neuronal population in which these proteins are expressed, and their gene expression is greatly important for the study of NRG/ErbB4 signaling. Here we determined an approximate proportion of 85% glutamatergic neurons and 15% GABAergic interneurons in hippocampal cultures, prepared from C57BL/6 mice in embryonic (E) day 18, similar to the proportion reported in vivo and in vitro. We also determined the expression of Nrg1 type III, Nrg2 and ErbB4 genes by the day in vitro (DIV) 7. Nrg1 type III had higher expression levels than Nrg2, similar to the reported in the hippocampus in vivo. Together, these results suggest that these cultures can be used as a valid model for the study of NRGs signaling in vitro.
Introducción
Las neuregulinas corresponden a una familia de factores de crecimiento y diferenciación, que sirven como ligandos para receptores tirosina quinasa de la familia ErbB (ErbB1-4). El gen Nrg1 codifica para múltiples isoformas (> 30), la mayoría de las cuales se expresan como proteínas transmembrana y se agrupan en seis tipos (I-VI), que difieren en los dominios N-terminal, siendo las NRG1 tipo I y III las principales en el cerebro. Al ser la NRG1 el miembro más estudiado de la familia, se conoce su importante participación como un regulador de la mielinización periférica, así como en gran variedad de funciones de regulación en sinapsis del Sistema Nervioso Central. Muchas de estas funciones son relevantes en el contexto de enfermedades neuropsiquiátricas, como la esquizofrenia (EZ) [1], [2].
La expresión de la NRG1 en el SNC es mayor antes del nacimiento, siendo la NRG1 tipo III la más abundante. Esta proteína se expresa en la mayoría de las neuronas glutamatérgicas, referidas también como neuronas de proyección o piramidales corticales, así como en un subconjunto de interneuronas GABAérgicas corticales y astrocitos [3], [4]. Por otra parte, su principal receptor en el cerebro, el ErbB4 se expresa prominentemente en distintas subclases de interneuronas corticales [5].
Otro miembro de esta familia de proteínas, que está relacionado con la NRG1, es la NRG2. El gen que codifica para esta proteína fue clonado basándose en la similitud de secuencia con el gen Nrg1 y su estructura proteica asemeja a la NRG1 tipo I [3]. De igual manera a esta, la proteína NRG2 sufre procesamientos proteolíticos que dan origen a su forma activa soluble y está involucrada en señalización paracrina hacia los receptores ErbB4 [6], [7]. A pesar del traslape en la especificidad de receptor, las NRG1 y NRG2 exhiben propiedades biológicas distintas en cultivos celulares [6]. Esto sugiere que ambas proteínas podrían provocar respuestas celulares distintas, mediante un único receptor.
La expresión de la NRG2 en el cerebro aumenta después del nacimiento, siendo más prominente en el compartimento somatodendrítico de las neuronas, incluyendo interneuronas GABAérgicas de la corteza, hipocampo y neuronas del cerebelo [7], [8]. Sin embargo, a pesar de que sus dominios de expresión en el SNC estén reportados, sus posibles funciones en el cerebro no han sido muy estudiadas.
Se han sugerido posibles configuraciones de señalización para las funciones de regulación mediadas por la NRG2, en sinapsis GABAérgicas y glutamatérgicas; señalización autocrina en interneuronas GABAérgicas [8] y bidireccional (de manera parcialmente independiente de ErbB4) en las células granulares del hipocampo [9]. Sin embargo, las vías de señalización corriente abajo no han sido investigadas. Por otro lado, se han reportado desregulación dopaminérgica y trastornos del comportamiento en ratones mutantes para la NRG2. Los anteriores traslapan con fenotipos descritos en los mutantes para el receptor ErbB4 [10], [11]. Esto también sugiere que la NRG2 actúa como ligando del receptor ErbB4 en el SNC, para modular procesos celulares de relevancia sináptica.
En diversas poblaciones, incluyendo a Costa Rica, se han asociado variantes de los genes que codifican para las proteínas NRG1 y ErbB4, a un mayor riesgo genético para manifestar EZ [12]–[17]. Por otra parte, la interacción de variantes de los genes Nrg1 y Nrg2, puede resultar en una mayor susceptibilidad a la EZ [18]. Esto implica que ocurren interacciones funcionales de estas proteínas durante los procesos de regulación, lo cual es relevante para el funcionamiento de la red neural. Sin embargo, todavía no está claro cómo esta interacción puede contribuir a una mayor susceptibilidad a la enfermedad.
Como un primer paso para el estudio de las funciones de señalización mediadas por la NRG2 en el contexto de otras NRGs in vitro, mediante cultivos primarios de neuronas de hipocampo murino, es necesario tener un marco conceptual con una caracterización del cultivo. Por lo anterior, en este trabajo se determinó el porcentaje de neuronas glutamatérgicas e interneuronas GABAérgicas, así como los niveles de expresión de Nrg1 tipo III, Nrg2, y ErbB4 en cultivos primarios, por análisis inmunocitoquímicos y PCR cuantitativo en tiempo real (qPCR), respectivamente.
Metodología
Animales
Se utilizaron ratones C57BL/6 mantenidos de acuerdo con protocolos debidamente aprobados por el Comité Institucional para el Cuidado y Uso de los Animales (CICUA-039-16) de la Universidad de Costa Rica (UCR) y las políticas animales del Instituto Max Planck de Medicina Experimental (MPI-em), aprobadas por el Estado de Baja Sajonia, Alemania.
Cultivo primario de neuronas embrionarias de hipocampo
Los cultivos de neuronas primarias de hipocampo fueron preparados a partir de ratones C57BL/6 en E18. Los hipocampos fueron aislados y lavados 2 veces con HBSS (BioWhittaker Cambrex, BE10-543F). Luego el tejido se disgregó enzimáticamente con solución de papaína (40 μl papaína (Sigma, LS003126), 10 μl DNaseI 10 mg/ml (Sigma, DN25) y 10 μl L-cisteína 24mg/ml (Sigma, C-7880) en 1 ml de medio Neurobasal (NB) (Gibco, 21103-049)) por 15 min a temperatura ambiente (TA). La papaína fue inactivada mediante dos lavados con medio NB suplementado (NB+) (2% suplemento B27 (Gibco, 17504-044), 1% PenStrep (Sigma, P0781), 1% Glutamax (Gibco, 35050-038)) más 10% de suero fetal bovino (SFB) (Gibco, 10106-169). El tejido fue triturado en 2 ml de medio NB+ más 5% SFB con suaves resuspensiones del medio de cultivo con una micropipeta P1000. Posteriormente, esto se filtró en un colador de cultivo celular (BD Falcon, 352340). Las neuronas fueron sembradas a una densidad de ~2.6x105 células/ pozo en placas de 12 pozos con cubreobjetos de 18 mm (previamente recubiertos con Poli-L-lisina (Sigma, P1274) diluida a 0.1 mg/ml en buffer borato), con 1 ml de medio NB+ más 5% SFB a 37°C y 5% CO2. Después de 24 h, se realizó cambio de medio por NB+ sin SFB. Cada 2 días se reemplazó un tercio del medio de cultivo por medio fresco sin SFB.
Extracción y Retrotranscripción de ARN
Se realizó la extracción de ARN de las neuronas primarias de hipocampo en DIV 7, utilizando el kit SV Total RNA Isolation System (Promega, Z3100), siguiendo las indicaciones del fabricante. Se utilizaron alrededor de ~2,1x106 células por tanda de extracción.
La síntesis de ADN complementario (ADNc) se realizó con el kit GoScriptTM Reverse Transcription System (Promega, A500) siguiendo las indicaciones del fabricante. Se utilizaron al menos 200 ng de ARN en 4 μl de H2Odd y 1 μl de primers al azar por cada reacción. El máster mix se preparó con: 4 μl de buffer de reacción GoScriptTM 5X, 4 μl de MgCl2 25 mM, 1 μl de mix de nucleótidos (0,5 mM de cada dNTP), 0.5 μl de RNasin, 1 μl de transcriptasa reversa GoScriptTM y 4,5 μl de H2Odd. La reacción de RT-PCR se realizó en termociclador (Analytik Jena) con el siguiente programa de termociclado: 70 °C 5 min, pausa, 25 °C 5 min, 42 °C 1 h, 70 °C 15 min, 4°C infinito. Las respectivas cuantificaciones se realizaron con el NanoDrop™ 2000 (ThermoFisher). Posteriormente, las muestras fueron almacenadas a -20 °C.
qPCR
Se realizó el análisis de expresión de los genes Nrg1 tipo III, Nrg2, ErbB4 utilizando el GoTaq® qPCR Máster Mix (Promega, A6001). Se utilizaron los primers: F5’-TTCTCACCACTGCTCCACAG-3’, R5’-CTCCACGTGTGGCTCTCAT-3’ (Nrg2); F5’-ACTCAGCCACAAACAACAGAAAC-3’, R5’- GAAGCACTCGCCTCCATT-3’ (Nrg1 tipo III); F5’-AGGAGTGAAATTGGACACAGC-3’, R5’-CCATCTTGGTACACAAACTGATTT-3’ (ErbB4); F5’-CTGCTCTTTCCCAGACGAGG-3’, R5’-AAGGCCACTTATCACCAGCC-3’ (β-actina).
Las reacciones se prepararon con: 4 μl ADNc (5 ng/μl), 0.1 μl primer forward (F) (10 pM), 0.1 μl primer reverse (R) (10 pM), 5 μl de máster mix GoTaq qPCR y 0,8 μl H2Odd. Las mismas se realizaron por cuadruplicado y se utilizó el equipo 7500 Fast Real-Time PCR System (Applied Biosystems) utilizando su protocolo estándar de cuantificación relativa.
La expresión relativa de los genes Nrg1 tipo III, Nrg2 y ErbB4 se presentó en unidades relativas de fluorescencia (rfu) utilizando los valores de 2-ΔΔCt. Se realizó una prueba T-Student con el programa GraphPad Prism 8 para determinar diferencias entre la expresión de los genes Nrg1 tipo III y Nrg2 al DIV 7.
Inmunotinción de neuronas primarias de hipocampo
El análisis inmunocitoquímico se realizó con neuronas primarias en DIV 8. Se eliminó el medio de cultivo a las células y se realizaron tres lavados con PBS 1X. Posteriormente se fijaron las células con PFA 4% durante 20 min, se realizaron dos lavados con PBS 1X y se permeabilizaron con metanol 100% (previamente enfriado a – 20) °C por 5 min.
Luego, se realizaron 3 lavados con PBS 1X y se realizó el bloqueo durante 1 h a TA con solución de bloqueo (2% suero fetal bovino ThermoFischer Scientific 10082139 2% suero de cabra Sigma Aldrich G9023, 2% gelatina de pescado Sigma Aldrich G7765, en PBS 1X). Se diluyeron los anticuerpos primarios GAD67 (Sigma, MAB5406; 1:1000) y Neurogranina (Sigma, 7-425; 1:250) en 10% de solución de bloqueo. Se realizó la incubación con anticuerpos primarios a 4 °C durante 24 h.
Posteriormente se realizaron tres lavados con PBS 1X y se diluyeron los anticuerpos secundarios Alexa 555 (Invitrogen, A21422; 1:500) y Alexa 488 (Invitrogen, A-11034; 1:100), junto con DAPI (ThermoFischer Scientific D1306; 1:2000) en solución de bloqueo al 5%. Se incubó por 1 h y 45 min a TA en oscuridad. Luego se realizaron tres lavados con PBS 1X y los cubreobjetos se montaron en portaobjetos con 1 gota de Aqua PolyMount (Polysciences, 18606-20), y se almacenaron a 4 °C.
Análisis de microscopía
Las imágenes se obtuvieron con el microscopio invertido de fluorescencia Zeiss Axio Observer Z1, con un aumento de 10X. Las fotografías fueron tomadas en cinco zonas previamente definidas para cada cubreobjetos. Se analizaron 20 fotografías por cultivo primario, utilizando el plugin Cell Counter del programa ImageJ (v. 1,52t). Se contabilizó la cantidad de neuronas positivas para el marcador neurogranina (Ng+) y para el marcador GAD67 (GAD67+).
Resultados
Proporción de neuronas glutamatérgicas y de interneuronas GABAérgicas en cultivos primarios de hipocampo murino
Los cultivos primarios de hipocampo murino son heterogéneos, estos están conformados principalmente por neuronas de glutamatérgicas e interneuronas GABAérgicas [3], [19]. Estas neuronas son de suma relevancia para estudios de la señalización de NRGs/ErbB4 [1].
Para analizar si los cultivos primarios realizados se asemejan a lo reportado en la literatura, se procedió a determinar la proporción de estos tipos neuronales mediante análisis inmunocitoquímicos. Se observaron células positivas para neurogranina (Ng+), indicando la presencia de neuronas glutamatérgicas y para GAD67 (GAD67+), lo cual confirmó la presencia de interneuronas GABAérgicas en los cultivos primarios de hipocampo, al DIV 8 (figura 1 A). En promedio, se determinó un porcentaje de 84,4% de neuronas de proyección y 15,6% de interneuronas en promedio, en dos cultivos independientes (figura 1 B). Por lo tanto, los resultados sugieren que la proporción de neuronas de proyección e interneuronas se mantiene constante entre distintos cultivos.
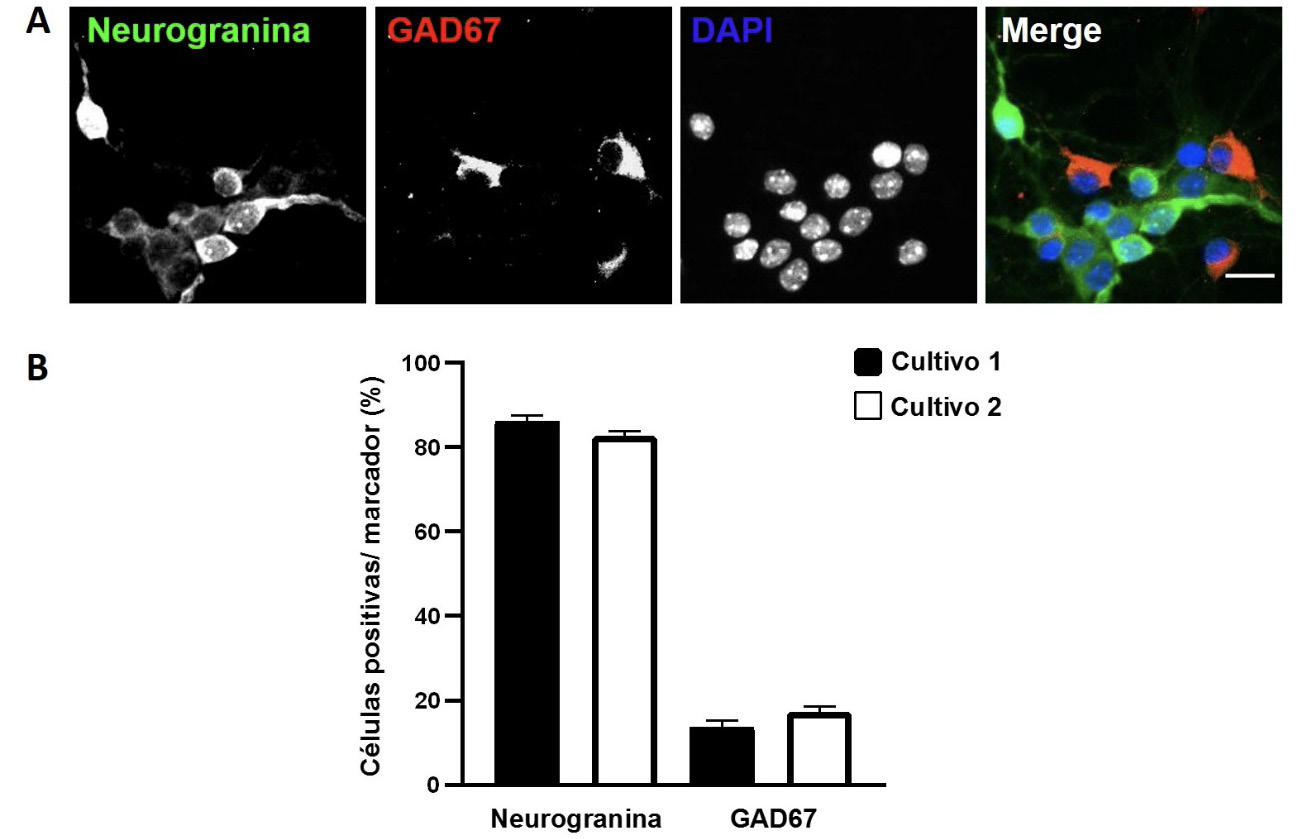
Figura 1. Proporción de neuronas glutamatérgicas e interneuronas GABAérgicas en dos cultivos primarios de hipocampo murino DIV 8 (A) Inmunotinción de neuronas primarias de hipocampo con Ng y GAD67. Barra de escala 25 μm. (B) Se determinó un porcentaje de 86,17% (±2,8) de células Ng+ y de 13,83% (±2,8) de células GAD67+ en el primer cultivo, y un 82,66% (±2,5) de células Ng+ y un 17.34% (±2,5) de células GAD67+ en el segundo cultivo. Los datos se muestran como promedio más SEM (error estándar).
Expresión de genes Nrg1 tipo III, Nrg2 y ErbB4 al DIV 7
Al igual que en condiciones in vivo, la expresión de genes en cultivos primarios es dinámica y puede verse afectada por diversos factores [20]. Por otro lado, se ha reportado la expresión de genes de interés para la señalización de NRGs en cultivos primarios de neuronas de hipocampo [3], [21], [22]. Sin embargo, esto podría variar entre distintos modelos, según por ejemplo su preparación. La expresión de Nrg1 tipo III, Nrg2 y ErbB4 fue analizada y se determinó que los tres genes se expresan al DIV 7, en tres cultivos primarios independientes. Por otro lado, se determinó que en estos cultivos el gen de Nrg1 tipo III muestra una mayor expresión, comparado con Nrg2 (figura 2).
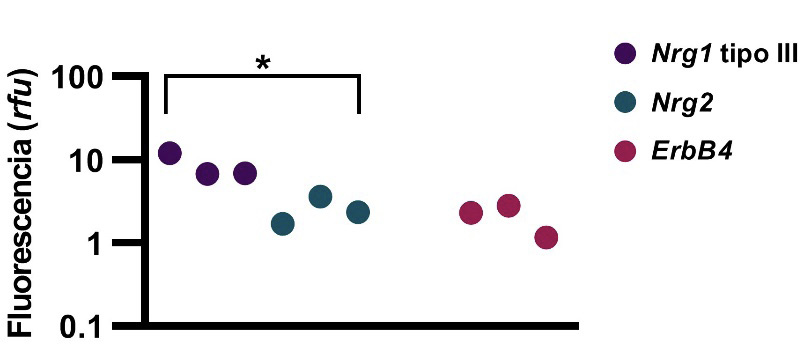
Figura 2. Expresión de Nrg1 tipo III, Nrg2 y ErbB4 en tres cultivos primarios de neuronas de hipocampo (E) 18 en DIV 7. Se determinó mayor expresión del gen Nrg1 tipo III mayor al gen Nrg2, T-Student, p = 0,0310, t=3,264, gl=4. Escala Log10.
Discusión
Los cultivos primarios neuronales son herramientas valiosas para el estudio de mecanismos regulatorios celulares, análisis bioquímicos y genéticos del SNC [23]. No obstante, presentan ciertas limitantes; particularmente la disminución de la alta heterogeneidad celular encontrada in vivo, la reducción en la complejidad de las interacciones celulares, la formación de circuitos aleatorios recurrentes y los posibles cambios en la expresión genética. Por lo que su caracterización es vital para validarlos como modelos de estudio [20], [24].
En el presente trabajo se determinaron aspectos importantes de los cultivos primarios de neuronas de hipocampo, relevantes en el contexto de algunas NRGs y ErbB4. Esta señalización es sumamente compleja, incluye un amplio número de ligandos y receptores distintos interactuando entre sí de manera altamente regulada para modular aspectos celulares a través de diversas vías de señalización celular, tanto en el neurodesarrollo como en la vida postnatal [1], [2], [25]. Los módulos de señalización de NRGs/ErbB4, involucran la interacción entre neuronas excitatorias glutamatérgicas e interneuronas GABAérgicas inhibitorias [2].
En la corteza in vivo, se ha reportado una proporción aproximada de 80% de neuronas glutamatérgicas y 20% de interneuronas GABAérgicas [4], [26]. También se ha observado esta proporción en cultivos in vitro preparados de hipocampo murino [19], [20]. Sin embargo, es necesario realizar una caracterización del cultivo que se pretende usar como modelo, para corroborar que se mantengan condiciones similares a las condiciones in vivo. De manera que, aunque no se tiene la conformación estructural, se pueda prever una composición que permita la formación de sinapsis excitatorias e inhibitorias y la interacción de módulos de señalización como es el caso de NRGs/ErbB4. El hecho de confirmar la presencia de ambos tipos neuronales y que su proporción concuerda a lo encontrado in vivo, sugiere que el modelo analizado es apto para el estudio in vitro de la señalización NRGs-ErbBs.
Por otro lado, se determinó la expresión de Nrg1 tipo III, Nrg2 y ErbB4 en los cultivos analizados. Esto concuerda con otros reportes, en donde se observa la expresión de los tres genes al realizar cultivos primarios de neuronas de hipocampo murino E 17-19 [3], [21], [22]. La expresión de ErbB4 ha sido demostrada en este DIV, y en el caso de Nrg1 tipo III, su expresión se ha evidenciado incluso desde el DIV 3[3], [27]. Lo anterior es muy relevante pues entre las limitaciones que pueden presentar los modelos in vitro, están los cambios en expresión génica [20], [24]. Por lo tanto, estos resultados sugieren que estos cultivos, son herramientas válidas y valiosas para el estudio de la señalización de NRGs in vitro.
En línea con lo anterior, la literatura reporta que la NRG1 tipo III se expresa mayoritariamente durante el desarrollo embrionario, mientras que la NRG2 aumenta su expresión durante la vida postnatal [7]. Los cultivos de neuronas primarias preparados en E18 y a los 7 DIV, que fueron analizados, presentan esa tendencia. Sería de interés realizar este análisis en varios DIV y con cultivos más maduros. De esta manera se tendría un mejor panorama del comportamiento de este modelo.
Agradecimientos
Se agradece al ITCR, al Instituto Max Planck de Medicina Experimental (MPI-em, Göttingen, Alemania), al CENIBiot y a Speratum S.A. por el apoyo, colaboración y por facilitar las instalaciones y equipos para el desarrollo de las metodologías de este trabajo. Se agradece al Programa de Pasantías para la Movilidad Estudiantil y a la Escuela de Agronegocios del ITCR, por la beca brindada a L.F.M.M. para realizar la pasantía en el MPI-em.
Referencias
[1] L. Mei and K. A. Nave, “Neuregulin-ERBB signaling in the nervous system and neuropsychiatric diseases,” Neuron, vol. 83, no. 1, pp. 27–49, 2014, doi: 10.1016/j.neuron.2014.06.007.
[2] L. Mei and W. C. Xiong, “Neuregulin 1 in neural development, synaptic plasticity and schizophrenia,” Nat. Rev. Neurosci., vol. 9, no. 6, pp. 437–452, 2008, doi: 10.1038/nrn2392.
[3] X. Liu et al., “Specific regulation of NRG1 isoform expression by neuronal activity,” J. Neurosci., vol. 31, no. 23, pp. 8491–8501, 2011, doi: 10.1523/JNEUROSCI.5317-10.2011.
[4] A. Agarwal, P. Dibaj, C. M. Kassmann, S. Goebbels, K. A. Nave, and M. H. Schwab, “In vivo imaging and noninvasive ablation of pyramidal neurons in adult NEX-CreERT2 mice,” Cereb. Cortex, vol. 22, no. 7, pp. 1473–1486, 2012, doi: 10.1093/cercor/bhr214.
[5] J. Neddens and A. Buonanno, “Selective populations of hippocampal interneurons express ErbB4 and their number and distribution is altered in ErbB4 knockout mice,” Hippocampus, vol. 20, no. 6, pp. 724–744, 2010, doi: 10.1002/hipo.20675.
[6] C. S. Crovello, C. Lai, L. C. Cantley, and K. L. Carraway, “Differential signaling by the epidermal growth factor-like growth factors neuregulin-1 and neuregulin-2,” J. Biol. Chem., vol. 273, no. 41, pp. 26954–26961, 1998, doi: 10.1074/jbc.273.41.26954.
[7] M. Longart, Y. Liu, I. Karavanova, and A. Buonanno, “Neuregulin-2 is Developmentally Regulated and Targeted to Dendrites of Central Neurons,” J. Comp. Neurol., vol. 472, no. 2, pp. 156–172, 2004, doi: 10.1002/cne.20016.
[8] D. Vullhorst et al., “A negative feedback loop controls NMDA receptor function in cortical interneurons via neuregulin 2/ErbB4 signalling,” Nat. Commun., vol. 6, no. June, 2015, doi: 10.1038/ncomms8222.
[9] K. H. Lee et al., “Bidirectional signaling of neuregulin-2 mediates formation of GABAergic synapses and maturation of glutamatergic synapses in newborn granule cells of postnatal hippocampus,” J. Neurosci., vol. 35, no. 50, pp. 16479–16493, 2015, doi: 10.1523/JNEUROSCI.1585-15.2015.
[10] I. Del Pino et al., “Erbb4 Deletion from Fast-Spiking Interneurons Causes Schizophrenia-like Phenotypes,” Neuron, vol. 79, no. 6, pp. 1152–1168, 2013, doi: 10.1016/j.neuron.2013.07.010.
[11] L. Yan et al., “Neuregulin-2 ablation results in dopamine dysregulation and severe behavioral phenotypes relevant to psychiatric disorders,” Nat. Publ. Gr., vol. 23, no. 5, pp. 1233–1243, 2018, doi: 10.1038/mp.2017.22.
[12] L. Athanasiu, M. Mattingsdal, A. K. Kähler, and A. Brown, “Europe PMC Funders Group Gene variants associated with schizophrenia in a Norwegian genome-wide study are replicated in a large European cohort,” vol. 44, no. 12, pp. 748–753, 2011, doi: 10.1016/j.jpsychires.2010.02.002.Gene.
[13] D. Li, D. A. Collier, and L. He, “Meta-analysis shows strong positive association of the neuregulin 1 (NRG1) gene with schizophrenia,” Hum. Mol. Genet., vol. 15, no. 12, pp. 1995–2002, 2006, doi: 10.1093/hmg/ddl122.
[14] M. R. Munafò, D. L. Thiselton, T. G. Clark, and J. Flint, “Association of the NRG1 gene and schizophrenia: A meta-analysis,” Mol. Psychiatry, vol. 11, no. 6, pp. 539–546, 2006, doi: 10.1038/sj.mp.4001817.
[15] N. Norton et al., “Evidence that interaction between neuregulin 1 and its receptor erbB4 increases susceptibility to schizophrenia,” Am. J. Med. Genet. - Neuropsychiatr. Genet., vol. 141 B, no. 1, pp. 96–101, 2006, doi: 10.1002/ajmg.b.30236.
[16] A. Agarwal et al., “Dysregulated expression of neuregulin-1 by cortical pyramidal neurons disrupts synaptic plasticity,” Cell Rep., vol. 8, no. 4, pp. 1130–1145, 2014, doi: 10.1016/j.celrep.2014.07.026.
[17] C. Walss-Bass et al., “A Novel Missense Mutation in the Transmembrane Domain of Neuregulin 1 is Associated with Schizophrenia,” Biol. Psychiatry, vol. 60, no. 6, pp. 548–553, 2006, doi: 10.1016/j.biopsych.2006.03.017.
[18] I. Benzel et al., “Interactions among genes in the ErbB-Neuregulin signalling network are associated with increased susceptibility to schizophrenia,” Behav. Brain Funct., vol. 3, pp. 1–11, 2007, doi: 10.1186/1744-9081-3-31.
[19] D. Vullhorst and A. Buonanno, “NMDA Receptors Regulate Neuregulin 2 Binding to ER-PM Junctions and Ectodomain Release,” Mol. Neurobiol., vol. 56, no. 12, pp. 8345–8363, 2019, doi: 10.1007/s12035-019-01659-w.
[20] A. Garrido-García et al., “Neurogranin Expression Is Regulated by Synaptic Activity and Promotes Synaptogenesis in Cultured Hippocampal Neurons,” Mol. Neurobiol., vol. 56, no. 11, pp. 7321–7337, 2019, doi: 10.1007/s12035-019-1593-3.
[21] P. Fazzari et al., “Control of cortical GABA circuitry development by Nrg1 and ErbB4 signalling,” Nature, vol. 464, no. 7293, pp. 1376–1380, 2010, doi: 10.1038/nature08928.
[22] K. M. Gerecke, J. M. Wyss, and S. L. Carroll, “Neuregulin-1 B induces neurite extension and arborization in cultured hippocampal neurons,” vol. 27, pp. 379–393, 2004, doi: 10.1016/j.mcn.2004.08.001.
[23] T. Xu, P. Molnar, C. Gregory, M. Das, T. Boland, and J. J. Hickman, “Electrophysiological characterization of embryonic hippocampal neurons cultured in a 3D collagen hydrogel,” Biomaterials, vol. 30, no. 26, pp. 4377–4383, 2009, doi: 10.1016/j.biomaterials.2009.04.047.
[24] R. I. Freshney, Culture of Animal Cells: a manual of basic technique and specialized applications, 6th ed. United Kingdom: Wiley-Blackwell, 2010.
[25] D. L. Falls, “Neuregulins: Functions, forms, and signaling strategies,” EGF Recept. Fam. Biol. Mech. Role Cancer, vol. 284, pp. 15–31, 2003, doi: 10.1016/B978-012160281-9/50003-7.
[26] H. Markram, M. Toledo-Rodriguez, Y. Wang, A. Gupta, G. Silberberg, and C. Wu, “Interneurons of the neocortical inhibitory system,” Nat. Rev. Neurosci., vol. 5, no. 10, pp. 793–807, 2004, doi: 10.1038/nrn1519.
[27] J. G. Allison, P. M. Das, J. Ma, F. M. Inglis, and F. E. Jones, “The ERBB4 intracellular domain (4ICD) regulates NRG1-induced gene expression in hippocampal neurons,” Neurosci. Res., vol. 70, no. 2, pp. 155–163, 2011, doi: 10.1016/j.neures.2011.02.009.
1 Ingeniero en Biotecnología. Escuela de Biología. Instituto Tecnológico de Costa Rica. Correo electrónico: luisfemonge@estudiantec.cr  https://orcid.org/0000-0002-3835-4707
https://orcid.org/0000-0002-3835-4707
2 Profesora/ Investigadora. Centro de Investigación en Biotecnología (CIB). Escuela de Biología. Instituto Tecnológico de Costa Rica. Correo electrónico: masoto@itcr.ac.cr  https://orcid.org/0000-0003-2266-787X
https://orcid.org/0000-0003-2266-787X