Caracterización de bioinsumos producidos artesanalmente en Nicaragua
Characterización of artisanally bioinoculant produced in Nicaragua
Johana O´Connor-Mendoza1, Leandro Páramo-Aguilera2,
Griselda Martínez-Laguna3, Laura Guillén-Rodríguez4
Fecha de recepción: 17 de enero de 2020
Fecha de aprobación: 28 de abril de 2020
O´Connor-Mendoza, J; Páramo-Aguilera, L;
Martínez-Laguna, G; Guillén-Rodríguez, L. Caracterización de bioinsumos producidos artesanalmente en Nicaragua. Tecnología en Marcha. Vol. 34-2. Abril-Junio 2021. Pág . 109-121. https://doi.org/10.18845/tm.v34i2.4961
Palabras claves
Caracterización microbiológica; identificación molecular; tinción; biofertilizante; biocontrolador.
Resumen
Para la caracterización microbiológica y molecular se aislaron e identificaron microorganismos cultivables de 4 bioinsumos comerciales provenientes del occidente y norte de Nicaragua. Esto involucró la tipificación morfológica a través de: tinción Gram para bacterias y observación de esporas para hongos filamentosos. Se realizó la extracción y secuenciación del ADN (gen ADNr 16S – bacterias, región ITS1- ITS4 hongos filamentosos) para obtener los arboles filogenéticos. Se logró la identificación molecular de 28 de 30 microorganismos aislados (23 bacterias: 12 especies, 11 géneros; 5 hongos filamentosos: 4 especies, 1 género). Encontrándose en los Bioisumos zona norte: muestra TS: 5 bacterias (Bacillus pumilus, Bacillus thuringiensis, 2 Bacillus sp. y Stenotrophomonas sp.) y 3 hongos filamentosos (Monascus pupureus, Neosartorya glabra y Aspergillus flavus- reportado como patógeno para cultivos); muestra LS: 6 bacterias (Bacillus megaterium, Bacillus subtilis, Bacillus sp., 2 Stenotrophomonas sp. y Paenibacillus sp.); muestra LL: 3 bacterias (Bacillus Megaterium, Staphylococcus succinus y Bacillus sp.) y 2 hongos filamentosos (Byssochlamys nívea – reportado como contaminante en fruta procesada y otro no identificado). Zona de occidente, muestra DCL: 9 bacterias (2 Lysinibacillus macroides, Bacillus subtilis, Bacillus flexus, Bacillus cereus – patógeno para el ser humano, Agrobacterium tumefaciens, 2 Bacillus sp. y una Stenotrophomonas sp.) y 1 hongo levaduriforme no identificado molecularmente. Lo anterior muestra gran diversidad microbiana y la presencia de patógenos en los bioinsumos que son perjudiciales para la planta y el ser humano.
Keywords
Microbiological characterization; molecular characterization; staining; biocontrol; biofertilizer.
Abstract
To develop the microbiological and molecular characterization, cultivable microorganisms from four commercial biofertilizers from western and northern Nicaragua were isolated and identified. This involved morphological identification through: Gram staining for bacteria and observation of spores for filamentous fungi. In order to obtain the phylogenetic trees, DNA extraction and sequencing was performed (16s rDNA gene for bacteria and ITS1 region for fungi). The molecular identification of 28 out of 30 isolated microorganisms was achieved (23 bacteria: 12 species, 11 genus, 5 filamentous; fungi: 4 species and 1 genus). Found in the northern zone inoculant: TS sample: 5 bacteria (Bacillus pumilus, Bacillus thuringiensis, 2 Bacillus sp. and Stenotrophomonas sp.) and 3 filamentous fungi (Monascus pupureus, Neosartorya glabra and Aspergillus flavus- reported as pathogen for crops). LS sample: 6 bacteria (Bacillus megaterium, Bacillus subtilis, Bacillus sp., 2 Stenotrophomonas sp., and Paenibacillus sp.). LL sample: 3 bacteria (Bacillus Megaterium, Staphylococcus succinus and Bacillus sp.) and 2 filamentous fungi (Byssochlamys nivea - reported as contaminants in processed fruit and another that was not sequenced). Western zone, DCL sample: 9 bacteria (2 Lysinibacillus macroides, Bacillus subtilis, Bacillus flexus, Bacillus cereus - pathogenic for humans, Agrobacterium tumefaciens, 2 Bacillus sp. and 1 Stenotrophomonas sp.) and 1 yeast fungus, that was not sequenced. The above shows great microbial diversity and the presence of pathogens in biofertilizer that are harmful to the plant and the human beings.
Introducción
La excesiva dependencia de los fertilizantes químicos para producir más cultivos inevitablemente daña la ecología ambiental y la salud humana con gran severidad. A nivel mundial en general y en Nicaragua en particular, país predominantemente agrícola, para contrarrestar el efecto nocivo de estas prácticas, existe una nueva corriente que promueve el consumo y producción orgánica [1]. En el país, existen serias limitaciones en la oferta y desarrollo de bienes y servicios orientados a la producción orgánica, uno de los casos particulares son los bioinsumos (biofertilizantes y biocontroladores de plagas) producidos a nivel local, la mayoría de estos productos poseen un bajo nivel tecnológico, por lo que se desconoce las alternativas de biocontrol (parasitoides, presencia del tipo de microorganismos, entre otros), no se tiene estudios del espectro de funcionamiento de los mismos. Sumado a esto, las instituciones públicas que deberían regular este tipo de productos, desconocen el tema, por lo que estos bioinsumos no cuentan con certificación, ni están registrados según lo indican las regulaciones para la certificación orgánica [2].
La caracterización de microorganismos y su posterior selección por su capacidad de hacer disponibles los nutrientes y promover su crecimiento, pueden contribuir a un posterior desarrollo de biofertilizantes. Con miras a lograr dicho objetivo, este estudio se planteó como un paso inicial la identificación molecular de microrganismos presentes en bioinsumos producidos artesanalmente en Nicaragua.
Materiales y métodos
En una primera etapa del proyecto de producción de bioinoculantes dirigida específicamente a la búsqueda de Bacillus subtilis, se aisló la fracción cultivable e identificó algunos microorganismos de interés (7 microorganismos) presentes en bioinsumos artesanales procedentes de las zonas seleccionadas (I y II), para la producción de un bioinsecticida a base de B.subtilis [3]. El presente trabajo muestra la completa identificación de toda fracción cultivable aislada y la caracterización de los microorganismos aislados en los bioinoculantes comerciales.
Aislamiento de microorganismos de bioinsumos artesanales
Se recolectaron 4 bioinsumos comerciales en presentaciones líquidas y sólidas [3], producidos en dos zonas del país (I y II), en donde la utilización de estos productos ha tenido un tremendo auge y los productos contaban con una autorización oficial del organismo competente. Estos se transportaron al laboratorio de microbiología de las aguas del Programa de Investigación Estudios Nacionales y Servicios Ambientales (PIENSA) de la Universidad Nacional de Ingeniería, ubicado en el Recinto Universitario Simón Bolívar, en la Avenida Universitaria, Managua, Nicaragua. Localizada geográficamente en las coordenadas UTM 12.1435887-86.2675189. El aislamiento de microorganismos se realizó en medios de cultivo sólidos (Agar Papa Dextrosa (PDA), Agar Plate Count (PCA), Agar Nutritivo (AN)) a partir de muestras individuales, para esto se inoculó en superficie, por triplicado. Posteriormente se seleccionaron los diferentes tipos de colonias obtenidas y se sembraron por estrías para la obtención de cultivos puros. Para los bioinsumos sólidos se prepararon filtrados, por medio de la suspensión aséptica de 1 g de muestra de bioinsumos en 5 mL de agua destilada estéril en un tubo de ensayo; en cambio para los bioinsumos líquidos solamente se tomó una muestra de 5 mL, la cual fue colocada en un tubo de ensayo. Todos los tubos con las muestras se agitaron en vortex por 20 s; luego se tomó 0.1 mL con una micropipeta y se depositó en gotas sobre el medio de cultivo en la placa Petri, el inóculo se distribuyó sobre toda la superficie del medio usando una espátula de Digralsky previamente esterilizada, finalmente todas las placas se incubaron a 35°C por 24 horas para bacterias y de 7 a 14 días para hongos [4].
Pruebas morfológicas a los microrganismos seleccionados
Se realizaron a través de observaciones al microscopio de luz OLYMPUS CHT, para las bacterias se efectuaron pruebas de tinción de Gram a las 24 horas de incubación con el objetivo de inmersión 100x, la observación de esporas para hongos filamentosos con cinta adhesiva transparente utilizando un objetivo de 40x. Los microorganismos se conservaron usando el método de transferencia periódica, que consiste en trasladar el microorganismo a un medio de cultivo fresco y nutritivo a intervalos que aseguren la viabilidad del mismo [5]. La transferencia se realizó en medio LB para bacterias y PDA para hongos filamentosos [6, 7].
Identificación molecular de los microorganismos seleccionados
Para el caso de las bacterias se amplificó la subunidad pequeña (16S) del ADNr con los cebadores pA (5’ AGA GTT TGA TCC TGG CTCAG 3’) y pc5B (5’ TAC CTT GTT ACG ACT T 3) [8]. La identificación taxonómica de los aislamientos de hongos se realizó con base en la amplificación de las regiones ITS1 (5 TCC GTA GGT GAA CCT GCG G 3)-ITS4 (5’ TCC TCCGCT TAT TGA TAT GC 3’) (Internal Transcribed Space) [9], estos genes son una alternativa para buscar la información filogenética y taxonómica de microorganismos. La comparación de las secuencias del gen ADNr 16S permite establecer relaciones filogenéticas entre organismos procariotas, de forma similar la región ITS permite hacer lo mismo en eucariotas [10].
Los electroferogramas recibidos fueron corregidos visualmente usando el programa Bioedit v7.0.9 [11]. Para la comparación de las secuencias, se obtuvieron de la base de datos del GenBank del National Center for Biotechnology Information (NCBI) secuencias de las regiones estudiadas de hongos y bacterias relacionadas taxonómicamente con los microorganismos, utilizando la herramienta BLAST (Basic Local Aligment Search Tool) para comparar secuencias altamente similares, de cada secuencia parcial del gen 16S para bacterias e ITS para hongos [12]. Para la identificación final, se realizó un alineamiento múltiple con las cinco secuencias elegidas para cada secuencia en estudio, utilizando la aplicación Clustal W Multiple alignment versión 1.4 incluida en el menú Accesory Application del programa Bioedit v7.0.9 y se utilizaron los parámetros por defecto [10]. Se utilizó el programa MEGA 7.0.14 para la construcción de los árboles filogenéticos, usando el método Neighbor-Joining Tree, evaluando la confiabilidad haciendo bootstrapping de 1000 repeticiones, teniendo en cuenta que cada rama se deberá repetir como mínimo en el 50% de las veces [9, 12].
Resultados y discusión
Aislamiento de microrganismos de bioinsumos artesanales
De los trabajos de aislamiento, se logró obtener 30 aislados diferentes de colonias de microorganismos en las muestras de bioinsumos, figura 1. Esta publicación aborda por primera vez en nuestro país el contenido microbiano completo, que fue posible aislar e identificar a partir de muestras de bioinsumos que están siendo actualmente comercializados y producidos a nivel artesanal.

Figura 1. Distribución de bacterias y hongos aislados de Bioinsumos artesanales, por zona de origen (TS: Tisey Sólido; LS: Licoroy Sólido; LL: Licoroy Líquido; DCL: Del Campo Líquido).
Herrmann et al., [13] caracterizaron el contenido microbiano de 65 biofertilizantes comerciales fabricados en los Estados Unidos, el Reino Unido, Australia, Sudáfrica, Tailandia, Kenia y Argentina. Los resultados mostraron que la mayoría (alrededor del 64%) contenía una o varias cepas de contaminantes y solo el 37% de los productos podían considerarse “puros”. El 40% de los productos probados no contenía ninguna de las cepas declaradas, sino solo contaminantes. Estos resultados se relacionan con los obtenidos en la presente investigación, donde se obtuvo que el 75% de los bioinsumos analizados contienen al menos una cepa contaminante y solo un 25% están libres de patógenos (figura 1).
El presente trabajo concuerda con Cuervo [4], en la necesidad de investigaciones que desarrollen un conocimiento paralelo a la preservación de los ecosistemas con el uso de biofertilizantes, con el fin de generar productos comerciales, los cuales también cuenten con pruebas de eficacia y estabilidad utilizadas con el objetivo de reducir costos, intensificar las interacciones biológicas y benéficas de los procesos naturales, proteger la salud y el ambiente. El presente trabajo, va encaminado en tal sentido.
Pruebas morfológicas a los aislados bacterianos
Aun cuando la morfología celular dice poco sobre las relaciones filogenéticas, sigue siendo útil para la identificación bacteriana. Por ejemplo: la presencia de endosporas y su localización resulta de mucha utilidad en la identificación de bacilos esporulados. Es posible sacar conclusiones en relación con la morfología de una bacteria, examinando una lámina que fue sometida a un proceso de tinción diferencial [14]. Algunos de los resultados obtenidos mediante aplicación Gram a cultivos bacterianos, se muestran en la figura 2.

Figura 2. Morfología en placa y tinción de Gram de aislados bacterianos y su procedencia. Muestra 1: AI, BI; Muestra 2: CI, DII; Muestra 3: CIII, DIII, Muestra 4: AII, AIII, BII, BIII, CII y DI.
De la aplicación de las pruebas de tinción diferencial Gram a los cultivos bacterianos se obtuvieron los siguientes resultados: 22 bacterias de forma bacilar (15 Gram positivas y 7 Gram negativas) y 1 de forma cocal Gram positiva. En la figura 2, se detallan 12 bacterias que se lograron identificar hasta nivel de especie como una muestra representativa del material aislado. Se puede observar que 11 bacterias se clasifican como Gram positivas, 10 poseen forma bacilar (DCL4-31F, TS13-2111U, LS6-11G, LS5-11B, DCL1-21A, TS11-31, LL9-21, DCL1-11, DCL2-4111H, DCL12-111Q) y un coco (LL8-111M); también se observó presencia de un bacilo Gram negativo (DCL12-21L). Los aislados bacterianos obtenidos se conservaron en medio de cultivo fresco LB en placas Petri a 10°C, con siembras periódicas mensuales para su posterior identificación vía molecular.
Pruebas morfológicas a los aislados de hongos filamentosos
Los hongos representan la mayor diversidad eucariótica sobre la tierra y ellos están entre los principales degradadores en los ecosistemas. Se ha estimado, conservadoramente, que existen alrededor de 1.5 millones de especies de hongos [15]. Muchas especies de hongos son patógenos importantes para plantas y humanos. Los resultados de la caracterización morfológica de los aislados puros de una muestra representativa (No incluye todo lo aislado) de hongos filamentosos se reflejan en el cuadro 1.
Cuadro 1. Descripción de los resultados de una muestra representativa de la caracterización macro y microscópica de los hongos filamentosos aislados de muestras de bioinsumos artesanales.
|
Código |
Color anverso de placa con PDA |
Apariencia micelio |
Forma de la espora |
|
TS1-2111 |
Blanco con centros verdosos y zonas marrones |
Algodonoso |
Esférica |
|
TS111-2111 |
Blanco |
Aterciopelada |
Esférica |
|
TS111-1212 |
Verde |
Pulvorolenta |
Esférica |
|
LLVII |
Blanco |
Algodonosa |
Esférica |
Fuente: [3]
En la figura 3, se observa una muestra representativa hongos filamentosos obtenidos por medio de la caracterización macroscópica describiendo el color y apariencia de las conidias; y de la identificación de esporas a través de las observaciones al microscopio de luz; para su posterior comparación con las referencias utilizadas [16].
Zeballos [17], aisló y purificó cepas provenientes de cuatro biofertilizantes diferentes, a partir de la purificación se obtuvo siete cepas de microorganismos: cuatro cepas de bacterias, dos de levaduras y una de hongo filamentoso. La baja abundancia y diversidad de microrganismos posiblemente se dio por la presencia de algunos ingredientes de los biofertilizantes que influyen en el medio de crecimiento. Esto difiere con los resultados de este estudio, ya que la variedad microbiológica es muy rica, teniendo en cuenta que se aislaron un total de 6 hongos: 5 filamentosos y 1 levaduriforme. Esto puede deberse al tipo de sustrato que se ha utilizado en la preparación.
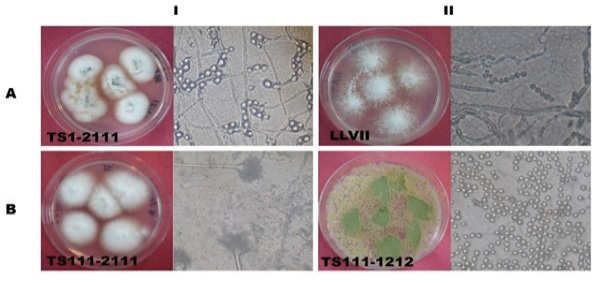
Figura 3. Morfología en placa y observación de esporas de algunos hongos filamentosos aislados de muestras de bioinsumos. AI, BI y BII: Muestra 1; AII: Muestra 3.
Identificación molecular de bacterias
El gen ribosomal 16S es frecuentemente empleado para estudios filogenéticos debido a que es una secuencia altamente conservada entre las diferentes especies de bacterias; el gen se encuentra ubicado en la subunidad pequeña del ribosoma (30S), asociado a esto, el gen ribosomal 16S es una región híper variable que prevé especificidad de especie, lo cual resulta muy útil para la identificación de especies bacterianas [12]. Los resultados obtenidos con las secuencias analizadas fueron utilizados para elaborar el árbol filogenético que se presenta en la figura 4.
De los 23 aislados de bacterias encontrados en los Bioinsumos, 12 se lograron identificar a nivel de especie mediante el análisis del árbol filogenético de la figura 4 y cuadro 2 como: TSII-31 (A1) como Bacillus thuringiensis, TS13-2111U(A3) como Bacillus Pumilus, LS5-11 (B) como Bacillus megaterium, DCL1-21(D1) como Lysinibacillus macroides, DCL12-111(D5) como Bacillus cereus, DCL1-11 (D6) como Lysinibacillus macroides, DCL4-31 (F) y LS6-11 (G) como Bacillus subtilis, DCL2-411 (H) como Bacillus flexus, DCL12-21 (L) como Agrobacterium tumefaciens, LL9-21(L1) como Bacillus megaterium, LL8-111 (M) como Staphylococcus succinus; 11 a nivel de género: TS13-1111J (A2), DCL3-4 (D4), LS5-21 (S1), LS6-211P(S3) como Stenotrophomonas sp.; TS13-3C (A4), DCL2-11(D2), DCL3-3 (D3), LL8-221 (L2), LS5-1K (S2), TS10-111(S5) como Bacillus sp. y LS7-2111(S4) como Paenibacillus sp. Cada especie bacteriana (cepa) confirma su identidad en su mayoría en 100% con las secuencias contenidas en el Genbank. De las 23 bacterias identificadas molecularmente, es importante destacar que 14 pertenecen al género Bacillus. A diferencia de lo obtenido por [4], el cual solo logró aislar e identificar únicamente tres especies microbianas (Bacillus subtilis en medio sólido, Bacillus firmus y Bacillus sphaericus en medio líquido) partiendo de dos presentaciones diferentes de biofertilizantes; el presente trabajo logró aislar e identificar un lote mucho mayor de bacterias (23), llegándose a identificar 12 a nivel de especie.

Figura 4. Se muestra el árbol filogenético de aislados bacterianos utilizando el método Neighbor-Joining con un Bootstrap de 1000 réplicas. El árbol fue realizado con el programa MEGA 7.0.14, el código de acceso al GenBank se especifica al inicio del nombre del microorganismo.
Cuadro 2. Se muestran los resultados de la identificación final de bacterias hasta el nivel de especie, mediante el análisis filogenético utilizando el árbol de la figura 4.
|
Código de secuencia |
Código de microorganismo |
Vecino cercano (Número de acceso) |
Máxima identidad (%) |
Identidad final |
|
A1 |
TSII-31 |
Bacillus thuringiensis (MH553093.1) |
99 |
Bacillus thuringiensis |
|
A3 |
TS13-2111U |
Bacillus Pumilus (MF692773.1) |
99 |
Bacillus Pumilus |
|
B |
LS5-11 |
Bacillus megaterium |
99 |
Bacillus Megaterium |
|
D1 |
DCL1-21(A) |
Lysinibacillus macroides (MH458746.1) |
100 |
Lysinibacillus macrolides |
|
D5 |
DCL12-111(Q) |
Bacillus cereus (MH915610.1) |
100 |
Bacillus cereus |
|
D6 |
DCL1-11 |
Lysinibacillus macroides (MF079282.1) |
100 |
Lysinibacillus macrolides |
|
F* |
DCL4-31 |
Bacillus subtilis |
99 |
Bacillus subtilis |
|
G* |
LS6-11 |
Bacillus subtilis |
99 |
Bacillus subtilis |
|
H* |
DCL2-4111 |
Bacillus flexus |
100 |
Bacillus flexus |
|
L* |
DCL12-21 |
Agrobacterium |
99 |
Agrobacterium tumefaciens |
|
L1 |
LL9-21 |
Bacillus megaterium (MH910311.1) |
100 |
Bacillus Megaterium |
|
M* |
LL8-111 |
Staphylococcus |
100 |
Staphylococcus succinus |
Nota: * microorganismos identificados en una primera etapa por [3]. En el cuadro se muestra el código de la secuencia, el código de laboratorio del microorganismo, identidad máxima con el vecino cercano y la identidad final de la bacteria.
Con el propósito de entender, la función desempeñada (Datos no mostrados) por los diferentes microorganismos que fueron aislados e identificados en el contexto de los bioinsumos en estudio, se analizó y se concluyó que empleando estos bioinsumos artesanales, en los cuales no se tiene control de los microorganismos que se producen y están presentes en el producto final; se estarían aplicando inóculos a los cultivos que pueden ser beneficiosos para algunos procesos de promoción del crecimiento vegetal (Bacillus Pumilus, Bacillus thuringiensis y Bacillus Subtilis), a la inhibición y resistencia de patógenos (Bacillus Pumilus, Bacillus thuringiensis, Bacillus cereus, Agrobacterium tumefaciens y Bacillus Subtilis). No obstante, estas propiedades benéficas no se presentan en todos los bioinsumos estudiados por igual y no es posible preservar esta propiedad benéfica en el tiempo y en cada lote de producción, debido a que el proceso de selección del material de partida es siempre aleatorio (suelo de bosques con materia orgánica en proceso de degradación). Adicionando además estiércol, melaza y suero láctico, con sus propias cargas microbianas [18].
Algunos de los bioinsumos estudiados, presentan bacterias patógenas al ser humano como Bacillus cereus y muchos otros pudieran estar desarrollando actividades que no son aplicables a todo tipo de suelos, ni a todo tipo de cultivos, por ejemplo: Agrobacterium tumefaciens y Staphylococcus succinus. Todos estos inconvenientes, se pretenden superar, con la selección de bacterias aisladas de estos bioinsumos, elegidas por la función que realicen en los cultivos, por su no patogenicidad para el ser humano, por no mostrar antagonismo e inhibición entre las mismas y por garantizar un número de unidades formadoras de colonia estable para el cultivo final en el bioproceso desarrollado para ello.
Identificación molecular de hongos
Los métodos moleculares como PCR agilizan la identificación de hongos analizando regiones específicas dentro de los genes. Según [19], las secuencias de espacios transcriptos internos (ITS) son de las más utilizadas en la identificación, como código de barras estándar para hongos. Para la identificación final de los hongos y corroborar los resultados morfológicos, se identificaron molecularmente a través de la secuenciación de la región ITS1-ITS4 (Internal Transcribed Space) del ADNr. De los resultados del análisis de las secuencias se obtuvo el siguiente árbol filogenético (figura 5). De los 6 hongos aislados de los bioinsumos artesanales en estudio, 4 de ellos se lograron identificar a nivel de especie: TSI-2III (B1) como Monascus purpureus, TSIII-2III (B3) como Neosartorya glabra, LLVII (B5) como Byssochlamys nivea, TSIII-1212 (B6) como Aspergillus flavus y 1 a nivel de género: TSII-2II (B3) como Neosartorya sp.

Figura 5. Árbol filogenético de hongos aislados de bioinsumos utilizando el método Neighbor-Joining con un Bootstrap de 1000 réplicas. El árbol fue realizado con el programa MEGA 7.0.14, el código de acceso al Gen Bank se especifica al inicio del nombre del microorganismo.
Estos resultados se resumen en el cuadro 3. Finalmente, uno de los hongos no se logró identificar debido a resultados no satisfactorios durante el proceso de secuenciación.
Cuadro 3. Se muestran los resultados de la identificación final de hongos aislados de bioinsumos. El cuadro resume el código de la secuencia y el código de laboratorio del microorganismo, identidad máxima con el vecino más cercano y la identidad final del hongo.
|
Código de secuencia |
Código de laboratorio del microorganismo |
Vecino cercano (Número de acceso) |
Máxima identidad (%) |
Identidad final |
|
B1 |
TSI-2III |
Monascus purpureus (KY828906.1) |
99 |
Monascus purpureus |
|
B2 |
TSII-2II |
Neosartorya sp. (KX953624.1) |
100 |
Neosartorya sp. |
|
B3 |
TSIII-2III |
Neosartorya glabra (KF815577.1) |
100 |
Neosartorya glabra |
|
B5 |
LLVII |
Byssochlamys nivea (MH860285.1) |
100 |
Byssochlamys nivea |
|
B6 |
TSIII-1212 |
Aspergillus flavus (MH864266.1) |
100 |
Aspergillus flavus |
El género Neosartorya (Familia Trichocomaceae) son hongos filamentosos caracteristicos del suelo y poseen diversas actividades y/o aplicaciones biotecnológicas (Datos no mostrados). El análisis de los resultados de la identificación de los hongos aislados de los bioinsumos (cuadro 3), nos muestra que algunos de los aislados identificados encuentran utilidad en la industria farmacéutica (Monascus purpureus y Neosartoya glabra [20], en procesos de biodegradación y en la agricultura (Neosartorya sp., Byssochlamys nívea y Neosartorya glabra [19,20] , algunos podrían resultar indeseables como (Byssochlamys nívea) y hasta algunos pudieran resultar patógenos para los seres humanos y los animales como Aspergillus flavus [21]. Lo anterior aunado al análisis hecho para las bacterias aisladas e identificadas, nos da una idea de cómo algunos de estos bioinsumos podrían estar afectando los cultivos mediante la introducción de hongos y bacterias que no son adecuados para todo tipo de cultivos y suelos, además de que estarían afectando la salud humana por esporas y aflatoxinas que producen diferentes enfermedades.
Un gran número de biofertilizantes comerciales están disponibles en todo el mundo; sin embargo, la calidad y la eficacia de muchos de ellos no están probadas ni aprobadas. En ausencia de biofertilizantes eficaces de buena calidad y calidad constante, no es probable que disminuya la dependencia del uso de fertilizantes minerales a corto plazo. Por lo tanto, la disponibilidad de biofertilizantes de alta calidad debe ser una prioridad, especialmente en países donde la producción agrícola juega un papel clave en la economía y la seguridad alimentaria.
Conclusiones
La realización de este trabajo permitió obtener aislados de la microbiota cultivable presente en las muestras de boinsumos analizadas, obteniéndose 30 aislados diferentes de colonias de microorganismos. Entre los aislados se obtuvo 23 bacterias y 7 hongos (6 filamentosos y 1 levaduriforme). De los aislados antes mencionados, se logró identificar el 93% de ellos. Identificándose de los aislados bacterianos (Bacillus thuringiensis, Bacillus Megaterium (2), Bacillus cereus, Lysinibacillus macroides, Bacillus subtilis (2), Bacillus flexus, Bacillus pumilus, Bacillus cereus, Agrobacterium tumefaciens, y Staphylococcus succinus). En cuanto a los hongos filamentosos aislados, se lograron identificar como: Monascus purpureus, Neosartorya glabra, Byssochlamys nivea, Aspergillus flavus, y Neosartorya sp.
El análisis de las aplicaciones de los aislados identificados, permitió determinar que el 75% de los bioinsumos analizados contienen al menos una cepa contaminante y solo un 25% contienen cepas no patógenas. El empleo de los bioinsumos estudiados podría ser beneficioso para algunos procesos de promoción del crecimiento vegetal mediante Bacillus Pumilus, Bacillus thuringiensis y Bacillus Subtilis, a la inhibición y resistencia de patógenos mediante Bacillus Pumilus, Bacillus thuringiensis, Bacillus cereus, Agrobacterium tumefaciens y Bacillus Subtilis. No obstante, estas propiedades benéficas no se presentan en todos los bioinsumos estudiados por igual y no es posible preservar esta propiedad benéfica en el tiempo y en cada lote de producción. Finalmente, algunos de estos bioinsumos presentan bacterias patógenas al ser humano como Bacillus cereus y muchos otros podrían estar desarrollando actividades que no son aplicables a todo tipo de suelos, ni a todo tipo de cultivos, por ejemplo: Agrobacterium tumefaciens y Staphylococcus succinus.
Agradecimientos
Los autores desean transmitir sus mayores muestras de gratitud a la dirección del programa PIENSA de la Universidad Nacional de Ingeniería, por facilitar las instalaciones de laboratorio para el desarrollo del presente trabajo y brindarnos todo el apoyo de sus técnicos para hacer realidad el presente trabajo. Al mismo tiempo transmitimos nuestro agradecimiento a la Rectoría de la Universidad Nacional de Ingeniería y al Consejo Nacional de Universidades, por el apoyo financiero brindado para la realización de esta investigación. A todos ellos, nuestra gratitud.
Referencias
[1] Mahanty, T., Bhattacharjee, S., Goswami, M. (2017). Biofertilizers: a potential approach for sustainable agriculture development. Environ Sci Pollut Res 24: 3315. https://doi.org/10.1007/s11356-016-8104-0.
[2] Kuhl, R. (2010). Caracterización de tecnologías y servicios necesarios para cerrar puntos críticos y brechas tecnológicas. Fundación para el Desarrollo Tecnológico Agropecuario y Forestal de Nicaragua (FUNICA).
[3] Méndez-Úbeda, J., M., Flores-Hernández, M., S y Páramo-Aguilera, L., A. (2017). Aislamiento e identificación de Bacillus subtilis y evaluación del antagonismo in vitro frente a hongos Fito patógenos. Rev. Cient.NEXO.30(2):96-110. ISSNE19959516.http://dx.doi.org/10.5377/nexo.v30i2.5530.http://revistas.uni.edu.ni/index.php/Nexo.
[4] Cuervo, J. (2010). Aislamiento y caracterización de Bacillus spp como fijadores biológicos de nitrógeno y solubilizadores de fosfatos en dos muestras de biofertilizantes comerciales. Trabajo de Grado para optar al título de Microbiólogo Agrícola y Veterinario, Universidad Nacional de Colombia, Bogotá.
[5] Hernández, D., & Loaiza, A. (2014). Selección de un método para la conservación y preservación de actinomicetos aislados del suelo del jardín botánico de la Universidad Tecnológica de Pereira (Trabajo de Grado). Universidad Tecnológica de Pereira, Pereira.
[6] Lennox, E.S. (1955). Transduction of Linked Genetic Characters of the Host by Bacteriophage P1. Virology. Vol.1, N°2, pp.190-206. DOI: 10.1016/0042-6822(55)90016-7. ISSN: 0042-6822.
[7] Atlas, R.M. (1993). Handbook of Microbiological Media, Parks L (ED.) CRC Press, INC.
[8] Kuske, C., R., Bams, S., M., and Busch, J., D. (1997). Diverse uncultivated bacterial groups from soils of the arid Southwestern United States that are present in many geographic regions. Appl Environ Microbiology. 63(9):3614-3621.
[9] Álvarez, C., Osorio, N., & Montoya, M. (2013). Identificación molecular de microorganismos asociados a la rizosfera de plantas de vainilla en Colombia. Acta Biológica Colombiana, 293-305.
[10] Rodríguez, C. (2013). Evaluación de microorganismos promotores de crecimiento vegetal en Tomate (Solanum lycopersicum) variedad Santa Clara, aislados de residuos Ligno Celulósicos de Higuerilla (Ricinus communis). Trabajo de grado presentado como requisito para optar al título de: Especialista en Microbiología Industrial, Universidad Católica de Manizalez, Colombia.
[11] Altschul, S.F., Gish, W., Miller, W., Myers, E.W and Lipman, D.J. (1990). Basic Local Alignment Search Tool. J. Mol. Biol. 215: 403-410
[12] Castillo-Reyes, F., Hernández-Castillo, F., Gallegos-Morales, G., Flores-Olivas, A., Rodríguez-Herrera, R., & Aguilar, C. (2015). Efectividad in vitro de Bacillus y polifenoles de plantas nativas de México sobre Rhizoctonia solani. Revista Mexicana de Ciencias Agrícolas, 549-562.
[13] Herrmann, L., Atieno, M., Bräu, L., Lesueur, D. (2015) Microbial Quality of Commercial Inoculants to Increase BNF and Nutrient Use Efficiency. Biological Nitrogen Fixation. DOI: 10.1002/9781119053095.ch101
[14] López-Jacomé, L., E., Hernández-Durán, M., H., Colín-Castro, C., A., Ortega-Peña, S., Cerón-González, G., y Franco-Cendejas, R. (2014). Las tinciones básicas en el laboratorio de microbiología. Investigación es Discapacidad. Vol. 3. Um, 1. pp 10-18. Disponible en: http://www.medigraphic.org.mx.
[15] Hawksworth, D., L. (2001). The magnitude of fungal diversity: the 1.5 million species estimate revisted. Mycological Research 105: 1422–1432.
[16] Arias, E., Piñeros, P. (2008). Aislamiento e identificación de hongos filamentosos de muestras de suelo de los páramos de Guasca y Cruz Verde. Trabajo de Grado para optar al título de Microbiólogas Industriales, Pontificia Universidad Javeriana, Bogotá.
[17] Zeballos, H., M., F., (2017). Caracterización de microorganismos de montaña (MM) en biofertilizantes artesanales. Tesis. Escuela Agrícola Panamericana, Zamorano. Honduras.
[18] Ito, S. (2006). Caracterización y evaluación de los factores que determinan la calidad nutricional e inocuidad en la producción de fertilizantes orgánicos fermentados. Tesis para optar a Magister Scientiae en Agricultura Ecológica. CATIE. Turrialba, Costa Rica.
[19] Fernández-Ortuño, D., Loza-Reyes, E. A., & Fraaije, B. (2010). The CYP51C gene, a reliable marker to resolve interspecific phylogenetic relationships within the Fusarium speces complex and a novel target for species-specific PCR. International Journal of Food Microbiology, (144), 301-309.
[20] Pinheiro, V., Desagiacomo, C., Michelin, M., Maller, A., Monteiro, L., Jorge, J., & Plizeli, M. (2017). Neosartorya glabra polygalacturonase produced from fruit peels as inducers has the potential for application in passion fruit and apple juices. Brazilian Journal of Food Technology, 20, e2016163. doi:10.1590/1981-6723.16316.
[21] Amaike, S., & Keller, N. (2011). Aspergillus flavus. The Annual Review of Phytopathology, 49, 108-133. doi: 10.1146/annurev-phyto-072910-095221.
1 Ing. Químico. Universidad Nacional de Ingeniería. Facultad de Ingeniería Química. FIQ. Nicaragua. Correo electrónico: johana.oconnor@fiq.uni.edu.ni https://orcid.org/0000-0002-1875-4578
2 PhD. Universidad Nacional de Ingeniería. Programa de Investigación, Estudios Nacionales y Servicios del Ambiente (PIENSA-UNI). Nicaragua. Autor para correspondencia. Correo electrónico: leandro.paramo@piensa.uni.edu.ni . https://orcid.org/0000-0002-3694-3698. Autor para correspondencia.
3 Ing.Químico. Universidad Nacional de Ingeniería. Facultad de Ingeniería Química. FIQ. Avenida Universitaria frente a la UCA. Managua, Nicaragua. Correo electrónico: gris3095@hotmail.com https://orcid.org/0000-0002-3455-4314
4 Ing.Químico. Universidad Nacional de Ingeniería. Facultad de Ingeniería Química. FIQ. Nicaragua. Correo electrónico: avangel07@yahoo.es https://orcid.org/0000-0003-0948-4331