Biología sintética en la obtención de compuestos de interés para la industria alimentaria
Synthetic biology as a tool used to obtain compounds of interest in the food industry
María Paula Ortuño-Fajardo1, Jose Rodolfo Chacón-Halabi2,
María Paula Flores-Espinoza3, Roger Aguilar-Bravo4
Fecha de recepción: 15 de noviembre de 2019
Fecha de aprobación: 28 de febrero de 2020
Ortuno-Fajardo, M.P; Chacón-Halabi, J. R;
Flores-Espinoza, M. P; Aguilar-Bravo, R. Biología sintética en la obtención de compuestos de interés para la industria alimentaria.Tecnología en Marcha. Vol. 34-1. Enero-Marzo 2021. Pág 69-79. https://doi.org/10.18845/tm.v34i1.4830
https://doi.org/10.18845/tm.v34i1.4830
Palabras clave
Biobrick; clonación modular; enzimas de restricción; metagenómica; biofábricas.
Resumen
La ingeniería de rutas metabólicas, por medio de la biología sintética, se ha convertido en una herramienta de suma importancia para la industria alimentaria, debido a que se busca la optimización de procesos y obtención de compuestos ya existentes. Es posible producir compuestos que son altamente codiciados por el mercado, como enzimas que participan en procesos de fermentación o síntesis de productos de alta demanda, o saborizantes obtenidos de forma natural a partir de microorganismos modificados. Dicha edición genética se ha logrado mediante las técnicas desarrolladas en la rama de la biología sintética a lo largo de los años, como, por ejemplo, el ensamblaje de constructos genéticos y de CRISPR y ARN, mensajeros sintéticos. Estas técnicas son utilizadas para aumentar la productividad de un compuesto de interés en un microorganismo. No obstante, es necesario tomar en cuenta que no todos los microorganismos tienen las herramientas genéticas para realizar las modificaciones post-traduccionales necesarias para otorgar funcionalidad a una enzima o proteína, y que con una ruta metabólica de novo pueden producirse también compuestos e intermediarios tóxicos para el hospedero elegido. Particularmente, en la industria alimentaria es común la elección de microorganismos huéspedes que se consideren seguros para el consumo humano (GRAS) como las bacterias ácido-lácticas (LAB) y, más recientemente, las levaduras del género Saccharomyces.
Keywords
Biobrick; modular cloning; restriction enzymes; biofactories; metagenomics.
Abstract
The engineering of metabolic pathways through synthetic biology has become an important tool for the food industry because it seeks the optimization of processes and production of compounds of interest. It is possible to produce compounds that are highly coveted, such as enzymes that take part in fermentation processes, or synthesis of products of high demand, or flavoring produced naturally by a microorganism. This can be achieved through genetic edition developed by synthetic biology through the assembly of genetic constructs CRISPR and synthetic mRNA. These techniques are used to increase the productivity of an organism that on its own produces a compound of interest, or to genetically modify a microorganism so that it can perform the synthesis of a product. However, it is necessary to take into account that not all microorganisms have the genetic tools necessary for the required post-translational modifications to activate enzyme or protein functions, and that the insertion of de novo pathways may result in both toxic compounds and intermediates for the chosen host. Particularly, it is common, when working in the food industry, to choose host microorganisms safe to be consumed by human beings, like GRAS organisms such as lactic acid bacteria and, more recently, yeasts of the Saccharomyces genus.
Introducción
La industria alimentaria es una de las ramas más grandes e importantes de la ciencia, debido a que se encarga de abastecer a prácticamente toda la población. Por esa razón es un campo en el que se buscan cada vez más innovaciones y métodos por medio de los cuales se puedan optimizar los procesos de obtención de compuestos importantes [1]. Es aquí donde entran herramientas como la biología sintética y la reingeniería de rutas metabólicas, para hacer la obtención de productos más eficiente, rápida y efectiva [2]. Sin embargo, el desarrollo de estos compuestos de interés se ha complicado debido a requerimientos más estrictos con las empresas, para que sus líneas de producción sean más responsables con el ambiente y reduzcan la huella de carbon, mientras que por parte de los consumidores, se mantiene la demanda de productos con mejor sabor y más nutritivos[3]. Por razones como estas es esencial construir rutas metabólicas de novo, para satisfacer la necesidad de compuestos cuya producción natural es menos eficiente que si se desarrollan de manera sintética. Llega incluso a resultar más viable y económicamente beneficioso fabricar compuestos a escala industrial, una vez que el proceso se ha optimizado para la producción masiva [4].
La presente actualización del estado del arte en materia de ingeniería de rutas metabólicas tiene como objetivos la comprensión de las herramientas fundamentales utilizadas en biología sintética para la producción de compuestos recombinantes de interés, así como del papel del microbioma y la generación de rutas metabólicas de novo para la producción de compuestos de valor comercial, especialmente para la industria de alimentos.
Biología sintética
La biología sintética es la rama de la biología que estudia la ingeniería o síntesis de sistemas complejos basados en mecanismos biológicos, para realizar procesos que no existen en la naturaleza, o mejorar los existentes [5]. Esta ciencia busca diseñar y construir modelos, componentes, redes y rutas biomoleculares para reprogramar microorganismos [6].
El término de ingeniería metabólica ha sido popular desde la década de los 80 y desde entonces el rango de compuestos que pueden sintetizarse se ha expandido, esto gracias a los avances que se han dado en áreas como la secuenciación de ADN, la creación de bases de datos de expresión de genes y reacciones metabólicas, y de herramientas para controlar estas rutas [7]. No obstante, no es la forma más maleable de producir debido a que hay que tomar en cuenta factores como costo y disponibilidad de sustratos, de catalización y del hospedero elegido; regulación genética y formas de maximizar la productividad del proceso y la purificación de compuestos [8]. Estos factores deben analizarse porque no todos los genes y, por ende, no todas las enzimas van a expresarse en todos los microorganismos que se tengan a disponibilidad; incluso, intermediarios que no son tóxicos para un organismo pueden serlo para otro [9]. Además, no todos los organismos poseen las mismas capacidades metabólicas para realizar modificaciones post-traduccionales esenciales para la expresión de algunas proteínas [10]. Se deben tomar en cuenta las regulaciones metabólicas propias con las que cuenta un organismo para que califique como candidato para la producción de un compuesto de interés [11].
A pesar de que existen sistemas de biología sintética para la caracterización de células y la edición genómica, generar células que producen un compuesto de interés sigue siendo un reto debido a las complejas interacciones metabólicas de regulación del metabolismo [12]. Por esta razón, se cuenta con metodologías que comprenden el diseño, la construcción y la prueba de estas rutas endógenas que se busca potenciar, o exógenas, para la generación de un compuesto ajeno al microorganismo seleccionado [12]. Dichas metodologías incluyen la obtención de bacterias rediseñadas (designer cells) por medio del acoplamiento de herramientas de edición genómica, síntesis química y ensamblaje de ADN.
Historia y fundamentos de la biología sintética
Desde la década de los 80, con la aparición de nuevas técnicas de edición genética y biología molecular, como la reacción en cadena de la polimerasa (PCR) y las herramientas de secuenciación, se ha buscado tomar provecho de estas conexiones circuitales bioquímicas [6]. La base de la biología sintética son fragmentos de ADN codificantes, comúnmente llamados biobricks, para características específicas, que pueden unirse entre sí para desarrollar circuitos más complejos [13]. Al unir varios biobricks, estos pueden introducirse en otros organismos, modificando sus genomas para inducirlos a producir un compuesto o inhibir una ruta metabólica que disminuye la productividad de un organismo.
La biología sintética ha seguido diferentes fases de desarrollo, identificadas de acuerdo con las tecnologías descubiertas y aplicadas en cada una. La primera de estas fases se dio en la década de 1970, cuando se comenzó a editar el genoma de microorganismos para producir compuestos de interés, como la insulina a partir de E. coli [14]. La segunda fase vio su luz con la creación de genomas sintéticos a partir de constructos artificiales ensamblados para que un microorganismo produjera proteínas completas características de otro [15]. La tercera y actual fase se concentra en la síntesis de genomas completos y posible creación de especies nuevas [17], lo que ha causado mucha discusión, ya que se argumenta que se podría perjudicar la herencia de ciertos genes, lo que podría resultar en agentes contaminantes y dañinos para la salud humana [16]. Estos avances han hecho de la biología sintética una herramienta cada vez más versátil para la producción de compuestos o productos útiles para mejorar la calidad de vida para los seres humanos y su entorno.
Las modificaciones que se realizan sobre el metabolismo de un microorganismo deben llevarse a cabo utilizando componentes clave para la edición génica, como lo son las enzimas específicas para catalizar las reacciones necesarias para la producción de un compuesto de interés. La actividad de dichas enzimas y cómo estas podrían actuar sobre el material genético y los procesos biológicos del organismo de interés pueden ser analizados por medio de la biología sintética y la bioinformática, gracias a gran cantidad de herramientas como bases de datos, mecanismos de secuenciación de ADN y reacciones metabólicas existentes [7]. Una de las herramientas primordiales son las enzimas específicas que se encargan de activar las reacciones para manipular las rutas metabólicas objetivo, velocidad de reacción y alta eficiencia, con lo que se consiguen mejores costos de producción debido a que estas encimas se necesitan en concentraciones menores con respecto a otras, dentro del metabolismo del microorganismo [18].
Los promotores juegan un rol de suma importancia en la biología sintética porque una gran parte del control de las rutas metabólicas biosintéticas está en la transcripción y el análisis de los transcriptos, por lo que los promotores (principalmente los inducibles), al iniciar la transcripción, funcionan como reguladores genéticos muy eficaces [18].
Principales técnicas utilizadas en la biología sintética
Ensamblaje de secuencias
En biología sintética y bioinformática, el ensamblaje de secuencias se refiere al alineamiento y mezcla de una gran cantidad de fragmentos de ADN, para la generación de secuencias de interés. Para realizar estos ensamblajes existen técnicas que permiten distintos niveles de eficiencia según sea necesario en cada proyecto.
Golden Gate
Este es un sistema que permite ensamblar múltiples fragmentos de ADN en una reacción. Se fundamenta en el uso de enzimas de restricción tipo IIS combinadas, como la BsaI con ADN ligasas, con el objetivo de ensamblar direccional y simultáneamente múltiples fragmentos de ADN (figura1). Con este método es posible obtener una alta eficiencia en la clonación porque el producto obtenido carece de un sitio de reconocimiento para las enzimas de restricción y se utiliza la incubación simple de múltiples constructos de entrada no digeridos y un vector, en presencia de enzimas de restricción y ligasas [19].
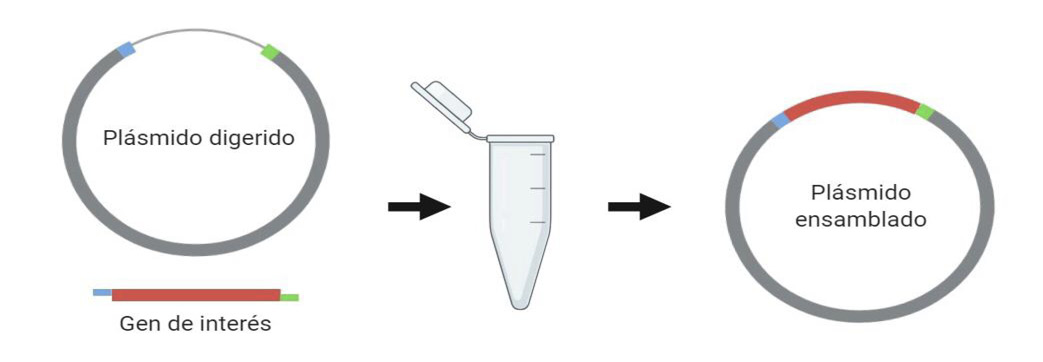
Figura 1. Sistema de ensamblaje Golden Gate. Para la utilización de este tipo de ensamblaje es necesaria la presencia de enzimas como BsaI y ADN ligasa. Generado por BioRender.com.
Gibson
El método de ensamblaje de Gibson permite la unión de forma fácil y rápida de largos constructos de ADN (fig.2). Dicha técnica involucra el uso de tres enzimas (T4 exonucleasa, la polimerasa de fusión y la Taq ADN ligasa) que llevan a cabo la ligación entre moléculas de ADN por traslape de secuencias. Se requiere la adición de extremos en las secuencias, denominados overhangs [20]. Para la creación de los overhangs, se utilizan primers de PCR entre biobricks adyacentes, de modo que los últimos 20 nucleótidos de una secuencia coincidan con los 20 nucleótidos iniciales de la siguiente. La T4 exonucleasa permite la adición de nucleótidos en los extremos de la secuencia, mientras que la Taq ligasa los une entre ellos [21].
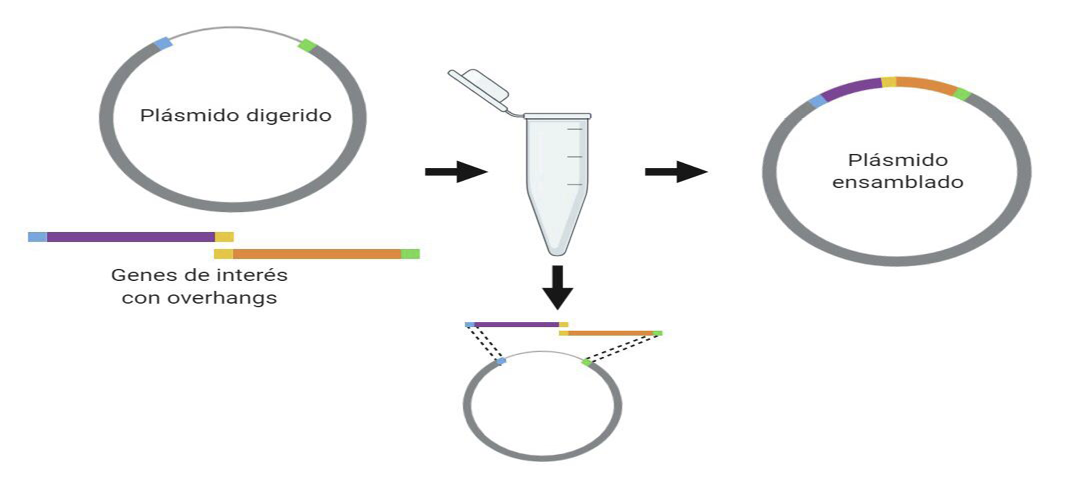
Figura 2. Sistema de ensamblaje Gibson. Este sistema emplea tres actividades enzimáticas distintas en un solo tubo de ensayo; la secuencia de fragmentos adjuntos es más larga que en el ensamblaje Golden Gate, lo cual favorece la obtención de porcentajes más altos de ensamblajes correctos. Generado por BioRender.com
Triple A (iGem)
El ensamblaje Triple A se aplica comúnmente en la biología sintética para la clasificación de unidades funcionales básicas de material genético (promotores, sitios de unión a ribosomas, secuencias de codificación de proteínas, terminadores, etc.) como “partes” que pueden ensamblarse en “dispositivos” o “sistemas” de nivel superior para realizar tareas complejas (fig.3). Al usar este método, una parte se designa como inserto y se digiere fuera de su plásmido usando las enzimas EcoRI y SpeI o XbaI y PstI [14]; la otra parte permanece en su plásmido, pero el plásmido se abre utilizando enzimas compatibles: EcoRI y XbaI o SpeI y PstI, respectivamente. Este protocolo requiere los siguientes pasos: 1) extracción de plásmidos que contienen las dos partes a ensamblar; 2) digestión de los plásmidos para crear extremos compatibles en fragmentos de ADN; 3) separación del ADN digerido por electroforesis en gel de agarosa; 4) extracción del fragmento seleccionado del gel; 5) ligadura de los fragmentos juntos, y 6) transformación del plásmido ligado en células [22].
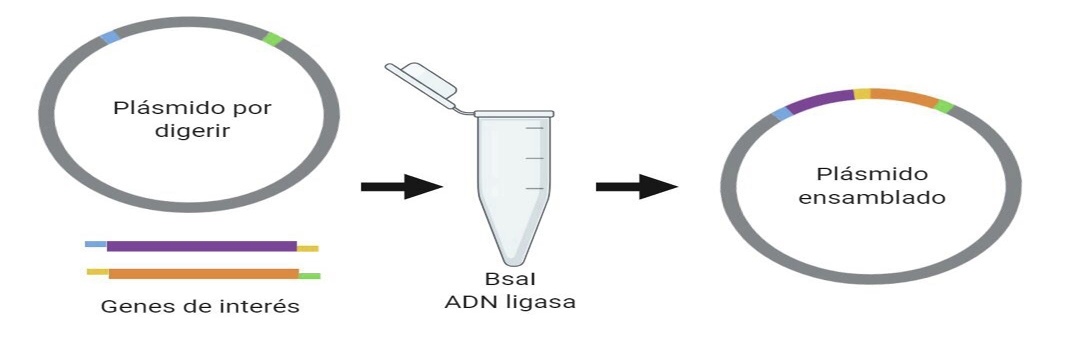
Figura 3. Sistema de ensamblaje Triple A. Por medio de herramientas bioinformáticas, se diseñan sitios de restricción en la cadena de ADN, que luego se digiere, con enzimas de restricción, para ligarse a un plásmido, utilizando ligasas;. Generado por BioRender.com
CRISPR
Llamado así por las siglas en inglés de Agrupación de Repeticiones Palindrómicas Regularmente Intercaladas. Es una herramienta utilizada en biología sintética para modificar genes de manera específica [23]. Esta técnica toma como base el sistema inmune utilizado por bacterias, debido a que cuando estos microorganismos son atacados por bacteriófagos que les insertan su ADN o ARN, unas enzimas llamadas Cas copian ese material genético en el sistema CRISPR/Cas9, y posteriormente utilizan esta información para reconocer la secuencia del material genético del virus y poder atacarlo [24].
De esta forma, el sistema CRISPR permite insertar genes por recombinación homóloga, lo cual se debe a su alta complementariedad con el blanco y la especificidad al inducir un corte en las cadenas de ADN para insertar una secuencia con alta homología, por los extremos 3’ y 5’ del sistema [26]. Con esta técnica, también se puede inducir una Reparación Dirigida por Homología (HDR), un método usado por las células para reparar lesiones en el ADN, con baja tasa de mutagénesis; asimismo, puede ser utilizada para llevar a cabo deleciones de fragmentos en ADN de doble cadena y utilizarse para detectar y generar mutaciones específicas en animales [26] [25].
Las perspectivas que se tienen en el uso de este sistema se enfocan hacia la mayor fidelidad y la especificidad del mecanismo. También se están llevando a cabo investigaciones en genética dirigida, con la idea de alterar el genoma de especies o poblaciones completas que tienen reproducción sexual [24]. Además, es importante mencionar que algunas hipótesis señalan que debido a que las proteínas Cas9 se extraen de S. aureus y S. pyogenes (microorganismos que pueden infectar a humanos), existe la posibilidad de que se cree resistencia a CRISPR/Cas9 en los humanos y, por tanto, se buscan alternativas de Cas de organismos que no han estado en contacto con humanos [24] [25] [27].
ARN mensajeros sintéticos
Este es un sistema que se basa en la construcción de un ARN mensajero, que se caracteriza por tener una secuencia complementaria de un micro-ARN target, y que ese ARN sintetizado codifique para una proteína apoptótica [23]. Después de introducido, el ARNm sintético puede seguir dos vías: la primera es que con las células que tengan el micro-ARN complementario, habrá formación de ARN de doble cadena y será reconocido por el complejo silenciador inducido por ARN, RISC por sus siglas en inglés, y degradado por tanto se impedirá la expresión de la proteína apoptótica; la segunda vía, que se abre cuando no está presente el micro-ARN target en las células diana o blanco, no posibilita la formación de ARN de doble cadena y, por ende, sí se expresa la proteína apoptótica, lo cual dará como resultado que la célula se “suicide”. Por tanto, esta herramienta permite purificar células de un fenotipo de interés, en situaciones en las cuales no se cuente con un citómetro de flujo acoplado a un cell sorter, teniendo como ventaja que se mantiene su viabilidad [23].
Producción de compuestos de interés en la industria alimentaria mediante rutas metabólicas de novo
Las bacterias no son los únicos microorganismos tomados en cuenta a la hora de seleccionar un microorganismo para la producción de un compuesto de interés en la industria alimentaria; se pueden usar las levaduras del género Saccharomyces. Entre los compuestos de mayor interés que producir están las enzimas, debido a que son beneficiosas durante el procesamiento de comidas. La manipulación de estas macromoléculas catalíticas permite la creación de productos con propiedades específicas como solubilidad, emulsión, actividad antioxidante y digestibilidad, todas características codiciadas en los alimentos [6]. A pesar de que la mayoría de levaduras no tienen la certificación GRAS, generalmente reconocida como seguridad para el consumo, Saccharomyces cerevisiae sí se cataloga como tal y se utiliza como chasis para producir proteínas recombinantes de manera heteróloga, como las que se mencionan a continuación [28].
La capacidad de las amilasas de digerir almidón crudo y procesado se aprovecha en la preparación de siropes con altos contenidos de glucosa, en la fermentación de bebidas y en las industrias de azúcar de todo el mundo. Las celulasas se emplean en la producción de azúcares fermentables a partir de fuentes lignificadas de células, y recientemente en la extracción y clarificación de extractos de frutas y vegetales. Otras enzimas que tienen papeles importantes en la fermentación para la producción de bebidas alcohólicas son las glucanasas, debido a que facilitan el proceso y, además, porque mejoran las cualidades visuales y olfativas de las bebidas. Las insulinasas resultan de interés debido a que descomponen moléculas para ser utilizadas como fuentes de carbono en los alimentos fructo-oligosacáridos, altamente buscados por la industria debido a que son bajos en calorías y altos en fibra [6].
Además de las enzimas mencionadas anteriormente, se pueden producir lipasas de manera sintética usando levaduras. Estas se emplean como saborizantes en productos lácteos, bebidas alcohólicas, en las industrias cárnicaas, y funcionan para extender la vida útil de los alimentos. Otro grupo enzimático que puede producirse siguiendo estas vías corresponde a las proteasas, las cuales se emplean como ablandadores de carne y en la manufactura de productos lácteos como el queso [6].
El grupo de bacterias LAB es un grupo heterogéneo de bacterias gram-positivas con muchas similitudes en lo que respecta a su metabolismo, además de ser catalogadas como organismos GRAS [29]. Estas bacterias son utilizadas comúnmente en procesos de fermentación de alimentos para humanos y animales, por lo que en las últimas décadas han sido objeto de estudio en investigaciones genéticas y de reingeniería. El objetivo de estas investigaciones ha sido diseñar nuevas cepas para la producción de proteínas, enzimas y productos metabólicos de interés en la industria alimentaria [29].
La generación de mutaciones cromosómicas en microorganismos LAB se ha logrado mediante técnicas como CRISPR, tanto para silenciar genes como para reemplazar genes dentro de rutas metabólicas, siendo un requisito que no afecte a los demás genes para catalogar al microorganismo como de grado alimenticio [30]. En bacterias ácido-lácticas se logró clonar la expresión de dos genes de la fresa: una enzima alcohol acil transferasa (SAAT) y una linalool/nerolidol sintasa (FaNES), ambas involucradas en la producción de metabolitos secundarios que les dan sabor a las fresas y otras frutas como el mango [31]. Como resultado, es posible usar estos compuestos como saborizantes que son considerados naturales, altamente codiciados en el mercado, debido a que son producidos por un microorganismo vivo.
Papel del microbioma en la industria alimentaria
Las comunidades de microbioma, que corresponden a los microorganismos presentes en el cuerpo del ser humano, pueden resultar responsables por la textura y las características organolépticas de alimentos fermentados [32]. Sin embargo, la presencia de microorganismos no deseados en uno de estos procesos industriales de producción de alimentos, puede significar una alteración en la calidad de la comida: su pudrición y seguridad de ingestión [33].
Metagenómica y procesos de fermentación
La metagenómica se refiere a un área de estudio que permite la extracción y el análisis de información genómica de microbiota no cultivada, es decir, de una muestra tomada del ambiente [34]. Esto resulta ventajoso sobre los métodos genómicos regulares debido a que no hay necesidad de hacer un cultivo de los microorganismos bajo condiciones controladas en el laboratorio [34].
Esta rama tiene el potencial de proveer información relacionada con la actividad microbiana involucrada en la producción de alimentos: la metagenómica ayuda a comprender cómo se va a comportar una comunidad microbiana al ser expuesta a una nueva enzima o compuesto producido, mientras que la metatranscriptómica permite la identificación de los genes expresados en la producción de un alimento y el cambio de la función que cumplen estos genes con el tiempo [33]. Este ha sido el caso de varios estudios realizados en torno a las bacterias ácido-lácticas o LAB, por sus siglas en inglés, y su función con respecto a la textura y la saborización de productos lácteos como el queso; esto puede verse alterado cuando suceden cambios en la expresión de genes cuando el consumo de carbohidratos, la proteólisis y el catabolismo de aminoácidos se ven modificados para promover la producción de moléculas saborizantes [35].
Futuro de la biología sintética en la industria alimentaria
La biología sintética adquiere cada vez más fuerza e importancia en el mundo, debido a la aplicación de las tecnologías desarrolladas. El área por trabajar en un futuro inmediato es el desarrollo de bebidas, helados, chocolates o chips que se clasifiquen como saludables, debido a su menor contenido de grasa, carbohidratos y calorías y para satisfacer la continua demanda de la población [36].
La industria alimentaria y la generación de productos lácteos enfrentan una serie de retos para la implementación de la biología sintética en sistemas de producción de alta calidad. Las proteínas de origen sintético tienen el potencial de cambiar la generación de productos lácteos con un valor adicional benéfico para el consumidor, de forma accesible económicamente. A pesar de que es un área que atrae el interés de muchos investigadores, no es la única que tiene el potencial de otorgar a la población seguridad alimentaria [37].
Los avances en biología sintética en materia de plantas se han rezagado en comparación con los sistemas diseñados para bacterias, levaduras y mamíferos, que están revolucionando las industrias farmacéuticas y alimentarias [37]. Para poder llegar a este punto con la modificación sintética de plantas es necesario estandarizar los componentes fundamentales para la implementación de estrategias de control de la expresión genética y los procesos celulares [38]. Así, será posible comenzar a trabajar en el aumento del valor nutricional de productos agrícolas de alta demanda, construir metabolismos sintéticos para mejorar el crecimiento y desarrollo de las plantas y reducir el uso de fertilizantes al sintetizar nuevas relaciones de simbiosis para la generación de micorrizas, entre otros [39] [40].
Conclusión
La biología sintética tiene un enorme potencial en casi todas las áreas relacionadas con la industria alimentaria y la nutrición, y puede impactar directamente de forma positiva la vida cotidiana. Para lograrlo, es posible utilizar múltiples técnicas de ensamblaje como Golden Gate, Gibson, Triple A, y otras de modificación genética como CRISPR y ARNm sintéticos. Con estas herramientas, es posible generar rutas metabólicas de novo e incrementar la eficiencia de las ya existentes en un microorganismo, con la finalidad de obtener un compuesto de interés para la industria alimentaria. Las plataformas biotecnológicas, como lo son las herramientas bioinformáticas, hacen más eficiente el proceso de investigación e innovación para las distintas aplicaciones de la disciplina.
Referencias
[1] M. Parzanese, “Tecnologías para la industria alimentaria” Ultrasonidos, ficha, 19, 1-9, 2016.
[2] D. Jullesson, F. David, B. Pfleger, & J. Nielsen, “Impact of synthetic biology and metabolic engineering on industrial production of fine chemicals”, Biotechnology advances, 33(7), 1395-1402, 2015..
[3] M. Arevalo-Villena, A. Briones-Perez, M. R. Corbo, M. Sinigaglia & A. Bevilacqua, “Biotechnological application of yeasts in food science: Starter cultures, probiotics and enzyme production,” Journal of Applied Microbiology, 123(6), 1360–1372, 2017. doi:10.1111/jam.13548
[4] J. Becker & C. Wittmann, “Advanced biotechnology: Metabolically engineered cells for the bio‐based production of chemicals and fuels, materials, and health‐care products,” Angewandte Chemie International Edition, 54(11), 3328-3350, 2015.
[5] S. A. Benner & A. M. Sismour, “Synthetic biology,” Nature Reviews Genetics, 6(7), 533–543, 2005. doi:10.1038/nrg1637
[6] A. S. Khalil & J. J. Collins, “Synthetic biology: Applications come of age,” Nature Reviews Genetics, 11(5), 367, 2010. doi:10.1038/nrg2775
[7] J. D. Keasling, “Manufacturing molecules through metabolic engineering. science”, Sicence, 330(6009), 1355–1358, 2010. doi:10.1126/science.1193990
[8] M. J. Smanski, H. Zhou, J. Claesen, B. Shen, M. A. Fischbach, & C. A. Voigt, “Synthetic biology to access and expand nature’s chemical diversity,” Nature Reviews Microbiology, 14(3), 135, 2016.
[9] Y. Guo, J. Dong, T. Zhou, J. Auxillos, T. Li, W. Zhang, ..., & J. Lin, “YeastFab: The design and construction of standard biological parts for metabolic engineering in Saccharomyces cerevisiae,” Nucleic Acids Research, 43(13), e88-e88, 2015.
[10] J. A. Jones, V. R. Vernacchio, D. M. Lachance, M. Lebovich, L. Fu, A. N. Shirke,..., & M. A. Koffas, “ePathOptimize: A combinatorial approach for transcriptional balancing of metabolic pathways,” Scientific Reports, 5, 11301, 2015.
[11] P. Calero & P. I. Nikel, “Chasing bacterial chassis for metabolic engineering: A perspective review from classical to non-traditional microorganisms,” Microbial Biotechnology, 2018. doi:10.1111/1751-7915.13292
[12] J. Nielsen & J. D. Keasling, “Engineering cellular metabolism,” Cell, 164(6), 1185–1197, 2016. doi:10.1016/j.cell.2016.02.004
[13] R. P. Shetty, D. Endy, , & T. F. Knight, “Engineering BioBrick vectors from BioBrick parts,” Journal of Biological Engineering, 2(1), 5, 2008. doi:10.1186/1754-1611-2-5
[14] D. E. Cameron, C. J. Bashor, & J. J. Collins, “A brief history of synthetic biology,” Nature Reviews Microbiology, 12(5), 381–390, 2014. doi:10.1038/nrmicro3239
[15] E. Pennisi, “Synthetic genome brings new life to bacterium,” Science, 328(5981), 958–959, 2010. doi:10.1126/science.328.5981.958
[16] P. Marliere, “The farther, the safer: A manifesto for securely navigating synthetic species away from the old living world,” Systems and Synthetic Biology, 3(1-4), 77–84, 2009. doi:10.1007/s11693-009-9040-9
[17] S. F. Yuan & H. S. Alper, “Metabolic engineering of microbial cell factories for production of nutraceuticals,” Microbial Cell Factories, 18(1), 2019. doi:10.1186/s12934-019-1096-y
[18] J. D. Keasling, Metabolic Engineering, 14(3), 189–195, 2012. doi:10.1016/j.ymben.2012.01.004
[19] S. Werner, C. Engler, E. Weber, R. Gruetzner, & S. Marillonnet, “Fast track assembly of multigene constructs using Golden Gate cloning and the MoClo system,” Bioengineered, 3(1), 38-43, 2012. https://doi.org/10.4161/bbug.3.1.18223
[20] L. Li, W. Jiang, & Y. Lu, “A modified Gibson assembly method for cloning large DNA fragments with high GC contents,” Synthetic Metabolic Pathways, 203–209, 2018. doi:10.1007/978-1-4939-7295-1_13
[21] C. Engler & S. Marillonnet “Golden Gate cloning,” in: “DNA Cloning and Assembly Methods,” Methods in Molecular Biology (Methods and Protocols), vol 1116, S. Valla and R. Lale (Eds.) . Humana Press, Totowa, NJ, 2014.
[22] M. A. Speer & T. L. Richard, “Amplified insert assembly: An optimized approach to standard assembly of BioBrickTM genetic circuits,” Journal of Biological Engineering, 5(1), 17, 2011. doi:10.1186/1754-1611-5-17
[23] C. Giménez, L. Curti, & F. Pereyra, “Dos herramientas de la biología sintética,” Revista Hospital Italiano, Buenos Aires, 36(3), 124-128, sept. de 2016.
[24] M. Adli, “The CRISPR tool kit for genome editing and beyond,” Nature Communications, 9(1), 2018. doi:10.1038/s41467-018-04252-2.
[25] C. T. Charlesworth et al. “Identification of pre-existing adaptive immunity to Cas9 proteins in humans,” BioArxhiv, 2018. doi.org/10.1101/243345.
[26] M. Lammoglia, R. Lozano, C. García, C. Avilez, V. Trejo, R. Muñoz, & C. López, “La revolución en ingeniería genética: Sistema CRISPR/Cas,” Investigación en Discapacidad, 5(2), 116-128, agosto de 2016.
[27] A. M Moreno. et al., “Exploring protein orthogonality in immune space: A case study with AAV and Cas9 orthologs.” Preprint at https://www.biorxiv.org/ content/early/2018/01/10/245985.
[28] K.J. Verstrepen, P.J. Chambers, I.S. Pretorius, A. Querol, & G.H. Fleet, The yeast handbook. Heidelberg: Springer, 2006.
[29] C. Peterbauer, T. Maischberger, & D. Haltrich, “Food-grade gene expression in lactic acid bacteria,” Biotechnology Journal, 6(9), 1147–1161, 2011. doi:10.1002/biot.201100034
[30] I. Hernandez, D. Molenaar, J. Beekwilder, H. Bouwmeester, & J. E. T. van Hylckama Vlieg, “Expression of plant flavor fenes in Lactococcus lactis,” Applied and Environmental Microbiology, 73(5), 1544–1552, 2007. doi:10.1128/aem.01870-06
[31] I. Hernández, D. Molenaar, J. Beekwilder, H. Bouwmeester, & J. E. van Hylckama Vlieg, “Expression of plant flavor genes in Lactococcus lactis,” Applied and Environmental Microbiology, 73(5), 1544-1552, 2007.
[32] P. Amon & I. Sanderson, “What is the microbiome?”, Archives of Disease in Childhood. Education & Practice Edition, 102(5), 257–260, 2017. doi:10.1136/archdischild-2016-311643
[33] F. De Filippis, E. Parente, & D. Ercolini, “Recent past, present, and future of the food microbiome,” Annual Review of Food Science and Technology, 9(1), 589–608, 2018. doi:10.1146/annurev-food-030117-012312
[34] S. Boddu & K. Divakar, “Metagenomics insight into environmental microbiome and their application in Food/Pharmaceutical Industry,” Microbial Biotechnology, vol. 2. Application in Food and Pharmacology, 2018. ISBN: 978-981-10-7139-3, 978-981-10-7140-9
[35] E. ,Dugat-Bony, C. Straub, A. Teissandier, D. Onésime, V. Loux, C. Monnet et al., “Overview of a Surface-Ripened Cheese Community Functioning by Meta-Omics Analyses,” PLoS ONE 10(4), e0124360, 2015. https://doi.org/10.1371/journal.pone.0124360
[36] A. Tyagi, A. Kumar, S. V. Aparna, R. H. Mallappa, S. Grover, & V. K. Batish, “Synthetic biology: Applications in the food sector,” Critical Reviews in Food Science and Nutrition, 56(11), 1777–1789, 2014. doi:10.1080/10408398.2013.782534
[37] C. Engler, M. Youles, R. Gruetzner, T. M. Ehnert, S. Werner, J. D. Jones,..., & S. Marillonnet, “A golden gate modular cloning toolbox for plants,” ACS Synthetic Biology, 3(11), 839-843, 2014.
[38] K. Müller, D. Siegel, F. R. Jahnke, K. Gerrer, S. Wend, E. L. Decker,..., & M. D. Zurbriggen, “A red light-controlled synthetic gene expression switch for plant systems,” Molecular BioSystems, 10(7), 1679-1688, 2014.
[39] C. Rogers, & G. E. Oldroyd, “Synthetic biology approaches to engineering the nitrogen symbiosis in cereals,”, Journal of Experimental Botany, 65(8), 1939-1946, 2014.
[40] M.S. Roell, & M. D. Zurbriggen, “The impact of synthetic biology for future agriculture and nutrition,” Current Opinion in Biotechnology, 61, 102–109, 2020. doi:10.1016/j.copbio.2019.10.004
1 Ingeniero en Biotecnología. Escuela de Biología, Instituto Tecnológico de Costa Rica, Sede Central. Costa Rica. Correo electrónico: mapaula.ortuno@gmail.com.  https://orcid.org/0000-0003-3825-4463
https://orcid.org/0000-0003-3825-4463
2 Ingeniero en Biotecnología. Escuela de Biología, Instituto Tecnológico de Costa Rica, Sede Central. Costa Rica. Correo electrónico: opochacon1711@gmail.com.  https://orcid.org/0000-0002-2087-3859
https://orcid.org/0000-0002-2087-3859
3 Ingeniero en Biotecnología. Escuela de Biología, Instituto Tecnológico de Costa Rica, Sede Central. Costa Rica. Correo electrónico: maria.olimpiadas92@gmail.com.  https://orcid.org/0000-0003-0478-4247
https://orcid.org/0000-0003-0478-4247
4 Ingeniero en Biotecnología. Escuela de Biología, Instituto Tecnológico de Costa Rica, Sede Central. Costa Rica. Correo electrónico: raguilarbravo25@gmail.com.  https://orcid.org/0000-0002-6842-3937
https://orcid.org/0000-0002-6842-3937