Análisis de ADN mediante espectroscopía Raman
utilizando el método SERS
Raman spectroscopy analysis of DNA using the SERS method
Stephanie Varela-Fonseca1, Ernesto Montero-Zeledón2, Laura Rojas- Rojas3, Alejandro Varela-Fonseca4, Dionisio Gutiérrez-Fallas5
Fecha de recepción: 3 de setiembre de 2018
Fecha de aceptación: 28 de diciembre de 2018
Varela-Fonseca, S; Montero-Zeledón, E; Rojas-Rojas, L; Varela-Fonseca, A; Gutiérrez-Fallas, D. Análisis de ADN mediante espectroscopía Raman utilizando el método SERS. Tecnología en Marcha. Vol. 32-3. Julio-Setiembre 2019. Pág. 118-125.
DOI: https://doi.org/10.18845/tm.v32i3.4501
Palabras clave
Espectroscopía Raman; SERS; nanopartículas; ADN.
Resumen
El avance tecnológico de las últimas décadas ha propiciado el desarrollo de nuevas metodologías para la caracterización de materiales de origen biológico. La espectroscopía Raman es utilizada principalmente en la caracterización de sustancias orgánicas e inorgánicas puras, sin embargo, recientemente se está utilizando para el estudio de organismos y estructuras moleculares de interés biológico. La principal dificultad en el análisis de estos materiales mediante la espectroscopía Raman es la fluorescencia que presentan las muestras al ser iluminadas con el haz de luz láser, pero mediante la técnica SERS (Surface Enhanced Raman Spectroscopy), es posible aumentar la señal Raman y reducir la interferencia de la fluorescencia. Para la aplicación de las técnicas SERS se pueden utilizar nanopartículas metálicas, principalmente oro y plata, de una adecuada distribución de tamaños. En este trabajo se presentan los resultados de la aplicación de técnica SERS a la caracterización espectroscópica del ADN de dos organismos diferentes, de un ser humano y de una bacteria del género Rickettsia, utilizando nanopartículas de oro y plata para la aplicación de la metodología.
Keywords
Raman spectroscopy; SERS; nanoparticles; DNA.
Abstract
The technological advances of the last decades have contributed to the development of new methodologies for the characterization of biological samples. Raman Spectroscopy has been used for the characterization of pure inorganic and organic compounds, but recently, it is being used to study molecular structures of biological importance. Fluorescence is usually present in Raman spectroscopy, shielding Raman signals, but using the SERS (Surface Enhanced Raman Spectroscopy) technique, it is possible to locally increase the Raman signal and to reduce the importance of the fluorescence effects on the samples. The SERS effect could be achieved by adding metallic nanoparticles, of a specific size distribution, to the sample. Usually gold and silver nanoparticles are used for SERS. In this research, DNA of human and Rickettsia bacteria were characterized by the SERS technique, with both silver and gold nanoparticles.
Introducción
Actualmente, la espectroscopía Raman está siendo utilizada en diversas áreas de la investigación científica, en las Ciencias Forenses, las Ciencias Biológicas, como también en la industria de polímeros y de productos farmacéuticos, entre otras. Esta técnica de espectroscopía vibracional se basa en la dispersión inelástica de luz monocromática, obtenida mediante luz de láser, normalmente en la región del espectro visible, infrarrojo y ultravioleta. La espectroscopía Raman permite la identificación estructural de las moléculas y puede ser utilizada en una gran variedad de materiales en sus distintas formas: películas delgadas, polvos, líquidos, muestras volumétricas e incluso gases. Para los análisis es suficiente una pequeña de cantidad de muestra (partículas micrométricas, volúmenes del orden de micro litros, superficies del orden de micrómetros) y en muchas ocasiones no se requiere una preparación especial de la muestra. Bajo las condiciones óptimas de medición, esta técnica no es destructiva.
En el área biológica, la espectroscopía Raman se ha utilizado en el análisis de células, tejido y microorganismos [1]. Específicamente, en el análisis de células bacterianas es posible obtener información estructural que permite identificar las células, e inclusive, el diferenciar células normales de células que presenten alguna patología, como el cáncer [2]. Además, mediante análisis más detallados, también se podría obtener información de la distribución espacial de las células en una muestra.
La espectroscopía Raman presenta un gran potencial como técnica de análisis complementario, para la detección de cambios estructurales en las células, inducido por enfermedades, como el cáncer o por efectos de los medicamentos o, inclusive, de los tratamientos, como es la exposición a la radiación ionizante [3]. Es por ello, que implementar y desarrollar las técnicas para el análisis de muestras biológicas en nuestro país mediante esta técnica podría contribuir al desarrollo de capacidades locales para al diagnóstico y el tratamiento de algunas enfermedades.
En este trabajo se presenta un procedimiento para obtener los espectros Raman de muestras de ADN mediante el método SERS (Surface Enhanced Raman Spectroscopy), en el cual se utilizan un conjunto de técnicas, frecuentemente necesarias, para el análisis de material de origen biológico.
SERS (Surface Enhanced Raman Spectroscopy)
El método SERS consiste en inducir un incremento de la señal de dispersión Raman de las moléculas que se encuentran en contacto con superficies metálicas rugosas o con nanoestructuras. Este incremento se explica mediante la resonancia de los electrones del metal con el campo eléctrico de la luz del láser incidente, actuando en forma localizada sobre las moléculas de la muestra en contacto o en regiones muy cercanas al metal [4]. Este efecto superficial es muy importante para el estudio de las muestras de material de origen biológico, pues de otro modo, la señal Raman sería muy débil y, además, suele superponerse con otros fenómenos de luminiscencia (como la fluorescencia), los cuales enmascaran las señales de interés, impidiendo obtener información del espectro [5]. Mediante la metodología SERS se puede incrementar la señal Raman en varios órdenes de magnitud, dependiendo de la naturaleza de la muestra, el metal utilizado y la longitud de onda y potencia del láser, entre otros [6].
Normalmente, los metales más utilizados para la aplicación de esta metodología son el oro y la plata, pues ambos aumentan considerablemente la intensidad de la señal Raman de la muestra. Sin embargo, frecuentemente se prefiere la preparación de substratos o nanopartículas de plata por su bajo costo, aún su menor estabilidad en el tiempo. En este trabajo se presentan los resultados de la utilización de suspensiones acuosas de nanopartículas de oro y plata, para la obtención de los espectros Raman de las muestras de ADN.
Materiales y métodos
Los espectros fueron obtenidos utilizando un microscopio Raman confocal, modelo DXR de la marca Thermo Scientific, con detector del tipo CCD, equipado con láseres de diodos con longitud de onda de 532 nm (Potencia máxima de 10 mW) y 780 nm (Pmáx= 24 mW) y un láser de gas de HeNe de 633 nm (Pmáx= 8 mW); con potencias que pueden variar en pasos de 0,1 mW hasta su valor máximo. Se utilizó el láser de 633 nm, con potencias de 0,2 mW y 0,3 mW, con 12 exposiciones y un tiempo de 5 s por cada una de ellas, pues fueron las condiciones que presentaron los mejores resultados.
Se prepararon suspensiones coloidales de nanopartículas de oro y de plata, utilizando el método de Lee y Meisel [7]. Los coloides de plata fueron sintetizados a partir de una disolución de nitrato de plata (AgNO3, Sigma-Aldrich) y citrato de sodio (Na3C6H5O7, J.T. Baker) como agente reductor. Se disolvieron 90 mg de AgNO3 en 500 ml de agua, llevándose lentamente, bajo agitación constante, hasta el punto de ebullición. Luego se añadieron 10 ml de una disolución de Na3C6H5O7 al 1%. La disolución se mantuvo en agitación constante a la temperatura del punto de ebullición, por un periodo de 50 min, obteniéndose una solución amarillo-verdosa. De esta solución se tomaron 50 ml (en tubos de laboratorio), se colocaron en centrifugación por un periodo de 30 min para eliminar el sobrenadante y finalmente se completó el volumen inicial nuevamente con agua de alta pureza. Mediante vibración ultrasónica se obtuvo una suspensión homogénea, para posteriormente ser almacenada a una temperatura de aproximadamente
4 °C.
Para la síntesis de nanopartículas de oro se utilizó 0,0242 g de ácido cloroáurico (HAuCl4) en 50 ml de agua Milli-Q y citrato de sodio como agente reductor y estabilizante. Bajo constante agitación y manteniendo la temperatura de la solución en ebullición, la disolución se tornó de color rojizo, retirándose de la plantilla caliente a los 40 min. Después, se centrifugaron las muestras de las nanopartículas, separando el sobrenadante, y se observó su distribución de tamaños mediante un microscopio electrónico de transmisión (TEM).
La efectividad de las nanopartículas de plata y oro se comprueba al obtener el espectro Raman en una disolución de 1,2-Di(4-Piridil) Etileno, en volúmenes iguales de 1 µl, antes de realizar los ensayos con las muestras de ADN [8].
Resultados y discusión
En la síntesis de las nanopartículas de plata y oro, usadas en la técnica SERS, se utilizaron dos metodologías reconocidas, lo cual permitió obtener las partículas requeridas para el método SERS. En la figura 1 se muestra la distribución de tamaños, la forma y la dispersión de las nanopartículas de plata y oro, sintetizadas con citrato de sodio como agente reductor. Las nanopartículas obtenidas son principalmente de geometría esférica, sin embargo, algunas tienen forma de barra. En la preparación de las nanopartículas la concentración de agente reductor y el tiempo de tratamiento se relacionan con el tamaño y la forma de las nanopartículas obtenidas [9]. Las nanopartículas sintetizadas con oro son más pequeñas que las de plata y no se encuentran tan aglomeradas (figura 1 c). Además, la variación de los diámetros es pequeña (figuras 1 c y d), con un promedio de 23,4 nm y desviación estándar de 4,2 nm. En contraste, las nanopartículas de plata están más aglomeradas, son más grandes, su diámetro promedio es 52,7 nm, y presentan una mayor variación (±12,4 nm).
Los espectros Raman del ADN muestran alta fluorescencia, sin embargo, con la técnica SERS es posible obtener información suficiente para caracterizar el material. En la figura 2 y la figura 3 se muestran los espectros de ADN de bacteria utilizando nanopartículas de plata. El espectro mostrado en la figura 2 tiene una señal Raman de alta intensidad cerca de 1000 cm-1; éste modo de oscilación es muy activo con las nanopartículas de plata, mientras que las bandas ubicadas entre 1200 cm-1 y 1600 cm-1, presentan una baja intensidad relativa. Por otra parte, el espectro Raman del ADN de bacteria, utilizando nanopartículas de oro, se muestra en la figura 3. En éste se aprecia que la intensidad absoluta de la señal Raman en 1000 cm-1 es menor, lo que provoca que las bandas vibracionales entre 1200 cm-1 y 1600 cm-1 muestren una mayor intensidad relativa.
El espectro Raman del ADN humano se muestra en las figuras 4 y 5 con nanopartículas de plata y oro, respectivamente. Se observa que el espectro obtenido con nanopartículas de oro presenta mucho más ruido y las frecuencias no están tan bien definidos como en los espectros obtenidos con nanopartículas de plata. Este fenómeno puede deberse a la relación entre el tamaño de las nanopartículas y la longitud de onda del láser, pues el diámetro de las nanopartículas de oro era aproximadamente la mitad del diámetro de las de plata. La señal Raman presenta una intensidad mayor cuando el diámetro de las nanopartículas es cercano a un décimo de la longitud de onda del láser utilizado [10].
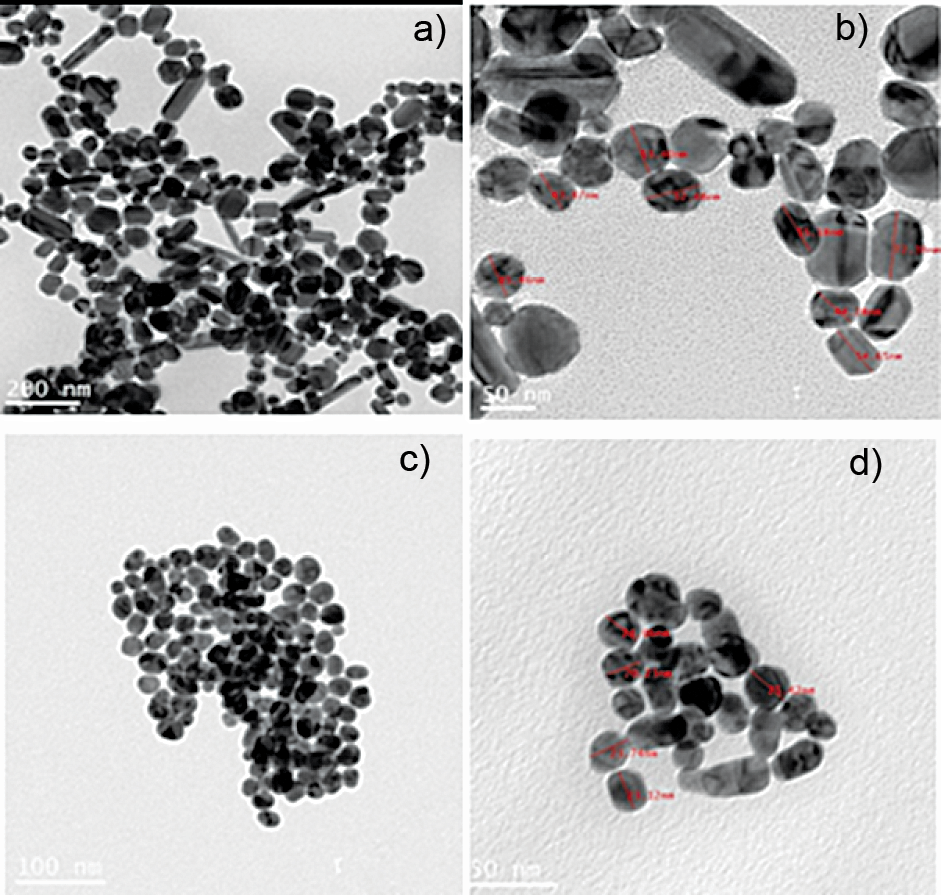
Figura 1. Micrografías TEM de las nanopartículas sintetizadas con citrato de sodio como agente reductor. En (a) y (b) se muestran las nanopartículas de plata y en (c) y (d) se muestran las nanopartículas de oro. (b) y (d) muestran algunas medidas de los diámetros encontrados.
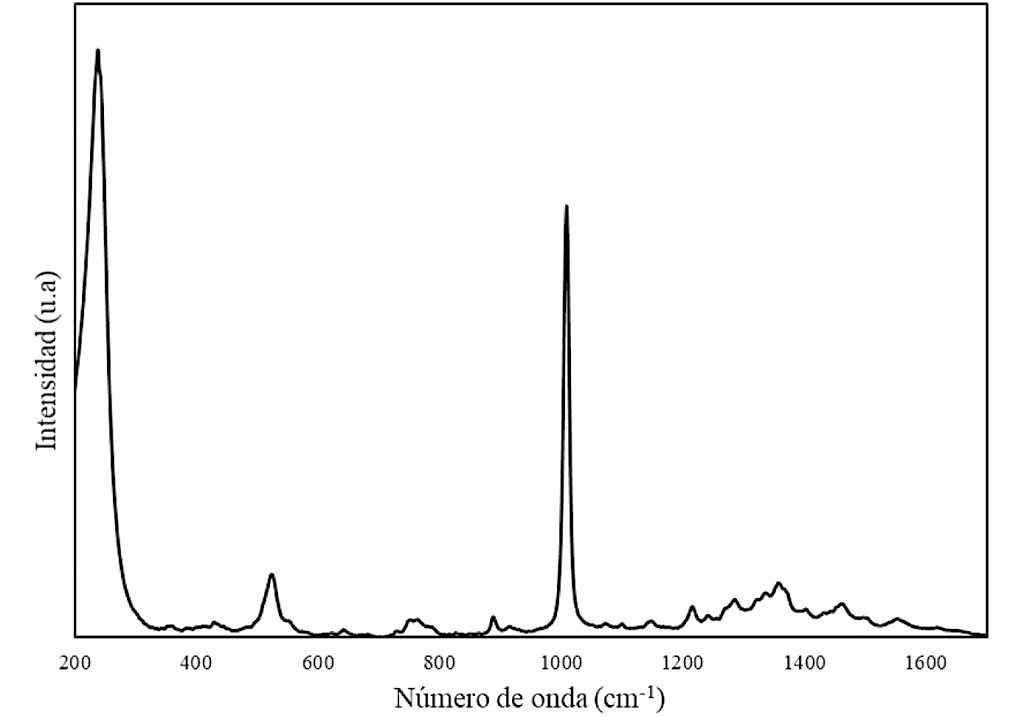
Figura 2. Espectro del ADN de bacteria obtenido con las nanopartículas de plata, sintetizadas con citrato de sodio como agente reductor
En las figuras 2, 3 y 4 se observa una banda Raman entre 520 cm-1 y 530 cm-1, que según Sundaram [11], está relacionada con los enlaces peptídicos y son atribuidas al estiramiento del enlace S-S, presente en las proteínas. También, en los espectros del ADN de bacteria se destaca una banda Raman en 1010 cm-1, atribuida a la lisina y fenilalanina [12]. Además, se observan otras vibraciones, como la banda de 1280 cm-1, que corresponde a amida III [11,12,13] pero también a fosfolípidos, proteínas, lípidos y a la timina [12]. Las vibraciones de la amida I, II se presentan en el rango de 1220 cm-1 - 1640 cm-1 [11]. Para Li [12] la señal Raman en 1369 cm-1, se debe al triptófano, porfirinas y lípidos, aunque también se observa para timina y proteínas [14]. Según Virkler y Lednev [15], alrededor de 1342 cm-1, la banda es debida al triptófano y al doblamiento del enlace C-H.
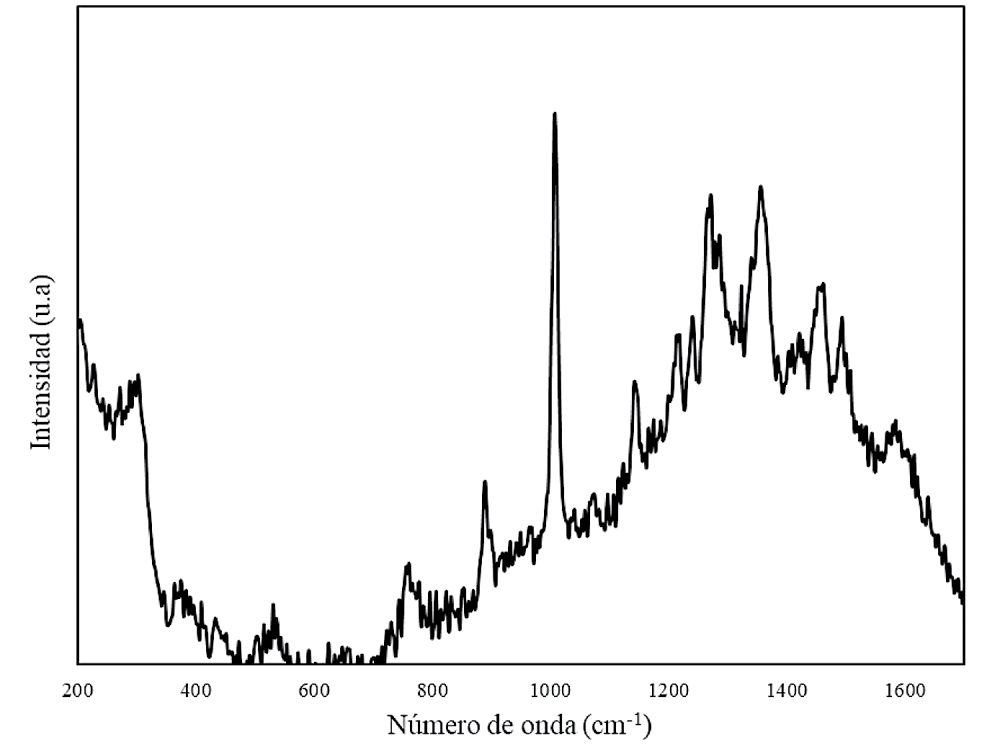
Figura 3. Espectro del ADN de bacteria obtenido con las nanopartículas de oro, sintetizadas con citrato de sodio como agente reductor
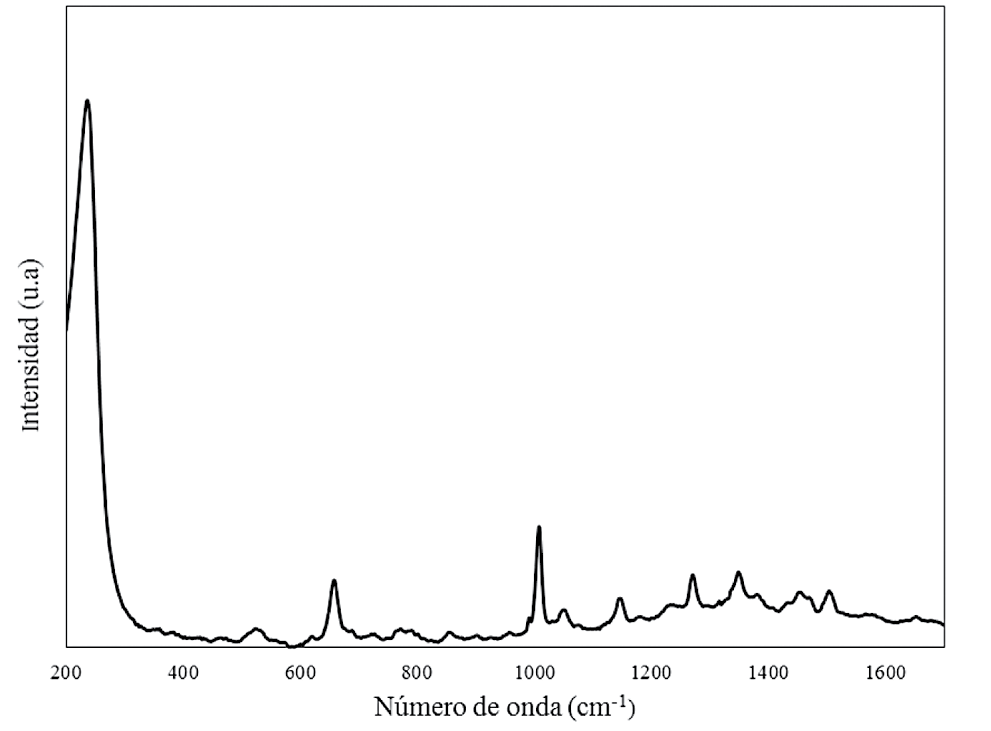
Figura 4. Espectro del ADN de humano obtenido con las nanopartículas de plata sintetizadas con citrato de sodio como agente reductor.
Una diferencia notoria entre los espectros Raman del ADN de bacterias y el humano está en la banda de 1010 cm-1, la cual es de mayor intensidad en los espectros de ADN de bacterias. Contrario a lo que ocurre con el ADN humano, pues a pesar de que la banda también está presente en algunos espectros, su intensidad es semejante a la intensidad de las bandas entre 1200 cm-1 y 1600 cm-1.
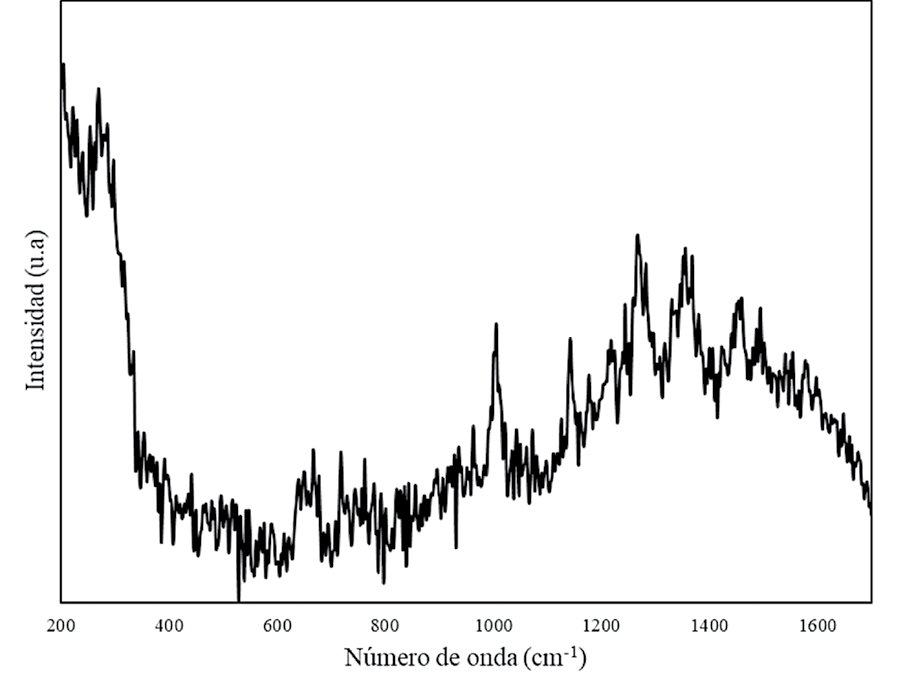
Figura 5. Espectro del ADN de humano obtenido con las nanopartículas de oro sintetizadas con citrato de sodio como agente reductor.
Conclusiones
Se han obtenido los espectros Raman de muestras de ADN mediante la técnica SERS. Los resultados permiten establecer el potencial del uso de esta técnica en el análisis e identificación de materiales de origen biológico. El análisis de los espectros obtenidos indica que las moléculas de ADN de bacterias y humanos presentan varias bandas definidas que podrían permitir su caracterización. Sin embargo, es necesario un análisis pormenorizado de estas señales, para lo cual también es necesario estudiar el efecto de tamaño, forma y tipo de metal de las nanopartículas, su relación con la estructura molecular y la intensidad de las señales Raman, la fluorescencia y resonancia en algunas frecuencias. Los resultados muestran la potencial aplicación de esta metodología en la caracterización de ADN, con la posibilidad de identificar los cambios estructurales que sean resultado de patologías u otro tipo alteraciones.
La experiencia y las capacidades para la síntesis de nanopartículas son fundamentales en el desarrollo de esta aplicación. Los resultados muestran que las metodologías estándares de síntesis de nanopartículas son adecuadas para las condiciones del Laboratorio de Espectroscopia Óptica de la Escuela de Física, sin embargo, existen métodos de síntesis que pueden facilitar el control de algunos de los factores, como es el tamaño y la forma de las nanopartículas.
Agradecimientos
Agradecemos el apoyo del Consejo de la Escuela de Física y el respaldo y financiamiento de la Vicerrectoría de Investigación y Extensión (VIE) del Instituto Tecnológico de Costa Rica. También agradecemos a la Escuela de Ciencia e Ingeniería de Materiales por el apoyo brindado para la realización de este proyecto y a la Facultad de Microbiología de la Universidad de Costa Rica, especialmente a Dihalá Picado Soto, por proveer el ADN que se utilizó en la preparación de las muestras.
Referencias
[1] R. Liu, D. Zhang, C. Cai, Y. Xiong, S. Li, Y. Su and M. Si, “NIR-SERS studies of DNA and DNA bases attached on polyvinyl alcohol (PVA) protected silver grass-like nanostructures”, Vibrational Spectroscopy, vol. 67, pp. 71-79, 2013.
[2] A. A. Bunaciu, H. Y. Aboul-Enein and V. D. Hoang, “Raman Spectroscopy for Protein Analysis”, Applied Spectroscopy Reviews, vol. 50, no. 5, pp. 377-386, 2015.
[3] M. Vendrell, K. Maiti, K. Dhaliwal and Y. Chang, “Surface-enhanced Raman scattering in cancer detection and imaging”, Trends in Biotechnology, vol. 31, no. 4, pp. 249-257, 2013.
[4] F. Gao, J. Lei and H. Ju, “Label-Free Surface-Enhanced Raman Spectroscopy for Sensitive DNA Detection by DNA-Mediated Silver Nanoparticle Growth”, Analytical Chemistry, vol. 85, no. 24, pp. 11788-11793, 2013.
[5] P. Chandra. “10.11.2 Optical Fibre”, Nanobiosensors for Personalized and Onsite Biomedical Diagnosis. Institution of Engineering and Technology, 2016.
[6] R. Aroca, Amplificación plasmónica de espectros Raman y de fluorescencia: SERS y SEF sobre nanoestructuras metálicas. Editorial CSIC Consejo Superior de Investigaciones Científicas, 2014.
[7] P. Lee and D. Meisel, “Adsorption and surface-enhanced Raman of dyes on silver and gold sols”, The Journal of Physical Chemistry, vol. 86, no. 17, pp. 3391-3395, 1982.
[8] A. Corzo, “Síntesis de Nanopartículas de Oro Obtenidas por Reducción de H[AuCl4]”, Rev Soc Quim Perú, pp. 79-79, 2012.
[9] P. L. Stiles, J. A. Dieringer, N. C. Shah y R. P. Van Duyne, “Surface-Enhanced Raman Spectroscopy,” Annual Review of Analytical Chemistry, vol. 1, p. 601–626, 2008.
[10] L. Qiu, P. Liu, L. Zhao, M. Wen, H. Yang, S. Fan and L. Zhou, “Analysis of plant genomic DNAs and the genetic relationship among plants by using surface-enhanced Raman spectroscopy”, Vibrational Spectroscopy, vol. 72, pp. 134-141, 2014.
[11] J. Sundaram, B. Park, A. Hinton, K. Lawrence and Y. Kwon, “Detection and differentiation of Salmonella serotypes using surface enhanced Raman scattering (SERS) technique”, Journal of Food Measurement and Characterization, vol. 7, no. 1, pp. 1-12, 2013.
[12] X. Li, T. Yang and J. Lin, “Spectral analysis of human saliva for detection of lung cancer using surface-enhanced Raman spectroscopy”, Journal Of Biomedical Optics, vol. 17, no. 3, p. 037003, 2012.
[13] R. Draga, M. Grimbergen, P. Viiverberg, C. Swol, T. Jonges, J. Kummer and J. Ruud Bosch, “In Vivo Bladder Cancer Diagnosis by High-Volume Raman Spectroscopy”, Analytical Chemistry, vol. 82, no. 14, pp. 5993-5999, 2010.
[14] C. Muntean, N. Leopold, C. Tripon, A. Coste and A. Halmagyi, “Surface-enhanced Raman spectroscopy of genomic DNA from in vitro grown tomato (Lycopersicon esculentum Mill.) cultivars before and after plant cryopreservation”, Spectrochimica Acta Part A: Molecular and Biomolecular Spectroscopy, vol. 144, pp. 107-114, 2015.
[15] K. Virkler and I. Lednev, “Raman spectroscopic signature of blood and its potential application to forensic body fluid identification”, Analytical And Bioanalytical Chemistry, vol. 396, no. 1, pp. 525-534, 2009.
1 Escuela de Ciencia e Ingeniería de los Materiales. Instituto Tecnológico de Costa Rica. Costa Rica. Correo electrónico: maruh.413@gmail.com. https://orcid.org/0000-0002-5613-5524
https://orcid.org/0000-0002-5613-5524
2 Escuela de Física. Instituto Tecnológico de Costa Rica. Costa Rica. Correo electrónico: emontero@tec.ac.cr.
3 Escuela de Física. Instituto Tecnológico de Costa Rica. Costa Rica. Correo electrónico: laurarojas@tec.ac.cr.
4 Escuela de Biología. Instituto Tecnológico de Costa Rica. Costa Rica.
5 Escuela de Física. Instituto Tecnológico de Costa Rica. Costa Rica. Correo electrónico: dgutierrez@tec.ac.cr.